Développement de packages R
Sophie Baillargeon, Université Laval
2021-03-29
- 1 Étape 1. Écrire les fonctions
- 2 Étape 2. Créer l’arborescence de répertoires et fichiers du package
- 3 Étape 3. Écrire la documentation des fonctions et du package
- 4 Étape 4. Vérifier et construire le package
- 5 Étape 5. Si désiré, partager le package
- 6 Étape 6. Au besoin, mettre à jour le package
- 7 Résumé
- Références
- Annexe
Note préliminaire : Lors de leur dernière mise à jour, ces notes ont été révisées en utilisant R version 4.0.3, le package devtools version 2.3.2, le package usethis version 2.0.1, le package roxygen2 version 7.1.1 et le package pkgbuild version 1.2.0. Le package ggplot2 version 3.3.3 aussi est utilisé dans un exemple. Pour d’autres versions, les informations peuvent différer.
Les packages R sont le meilleur moyen de partager des fonctions R. Pour créer un package R, il faut d’abord développer une arborescence de répertoires et fichiers similaire à ce qui se retrouve dans n’importe quel package source. Pour voir de quoi a l’air une telle arborescence, il suffit de :
- télécharger un package source à partir du CRAN (par exemple ggplot2_3.3.3.tar.gz sur http://CRAN.R-project.org/package=ggplot2),
- décompresser le fichier (il y a 2 étapes de décompression à effectuer).
L’exemple utilisé ici, soit le package ggplot2, est un gros package. L’arborescence de répertoires et fichiers formant son package source peut être représentée schématiquement comme suit :
ggplot2/
|-- build/
|-- data/
|-- inst/
|-- man/
|-- R/
|-- tests/
|-- vignettes/
|-- DESCRIPTION
|-- LICENSE
|-- MD5
|-- NAMESPACE
|-- NEWS.md
|-- README.mdSeuls certains de ces répertoires (dénotés par le caractère / en suffixe) et fichiers sont nécessaires dans tout package R. Certains fichiers, tels que DESCRIPTION et NAMESPACE, sont obligatoires et doivent suivre une syntaxe particulière.
Après avoir développé les fichiers source, qui incluent le code source et de la documentation, il ne reste plus qu’à construire l’archive (fichier compressé) du package. Nous verrons ici comment réaliser toutes ces étapes.
1 Étape 1. Écrire les fonctions
La première étape, celle de l’écriture des fonctions, fait appel à ce que nous avons appris dans toutes les notes de cours précédentes, en particulier celles sur les fonctions en R.
Lorsque nous créons des fonctions dans le but de les inclure dans un package, il faut garder en tête le chemin de recherche complet qu’utilisera R lors de l’exécution de ces fonctions. Ce chemin est le suivant :
- l’environnement local d’exécution de la fonction,
- l’environnement englobant de la fonction, soit l’espace de noms du package,
- l’environnement des objets importés par le package,
- l’environnement du package R de base,
- l’environnement de travail,
- les environnements de tous les packages chargés.
Nous savons déjà que, dans ce chemin de recherche, nous ne pouvons pas nous fier au contenu de l’environnement de travail, car il varie constamment en cours de session. Nous ne pouvons pas non plus nous fier aux packages chargés. En effet, la liste des packages chargés varie aussi en cours de session et d’un utilisateur à l’autre. Le bon fonctionnement des fonctions d’un package devrait dépendre d’un seul appel à la fonction library, celui servant à charger le package en question.
Ainsi, dans le corps des fonctions d’un package, nous pouvons toujours faire appel
- aux arguments et variables locales,
- à tout objet, public ou privé, contenu dans le package,
- aux objets provenant d’autres packages qui sont importés par le package,
- à des objets du package R de base, qui est toujours inclus dans le chemin de recherche.
Nous verrons comment inclure des objets provenant d’autres packages dans l’environnement des objets importés par le package. Dans le corps de nos fonctions, il est recommandé de faire appel à ces objets en utilisant l’opérateur :: pour indiquer clairement de quel package ils proviennent. Par exemple, si nous voulons utiliser la fonction dist du package stats, il est préférable de l’appeler comme suit : stats::dist().
Aussi, si notre but est éventuellement de rendre notre package public sur le CRAN, il faut respecter les politiques du CRAN, présentées ici : http://cran.r-project.org/web/packages/policies.html.
La plus importante de ces politiques est que notre package doit passer le R CMD check --as-cran sans erreurs ni avertissements (nous en reparlons plus loin). Pour passer cette vérification sans problèmes, notre code ne devrait pas, notamment :
- contenir des symboles non-ASCII (donc pas d’accents) :
afin de s’assurer que le code est fonctionnel sur n’importe quelle plateforme informatique; - utiliser d’association partielle d’argument (donc nous devons utiliser le nom complet des arguments dans les appels de fonction) :
afin de s’assurer que le code demeure fonctionnel si des arguments sont ajoutés aux définitions des fonctions appelées dans le code (ces nouveaux arguments pourraient porter des noms qui entrent en conflit avec l’association partielle); - toujours utiliser comme valeurs logiques
TRUEouFALSE(donc pas deTouF) :
parce qu’un objet R nomméTouFpeut être défini, écrasant du coup les définitionsT <- TRUEetF <- FALSE, alors qu’il est impossible de créer un nouvel objet R nomméTRUEouFALSE(mots-clés protégés); - contenir des exemples longs à rouler dans les fiches d’aide :
parce que les gestionnaires du CRAN roulent quotidiennement leR CMD check --as-cransur tous les packages du CRAN, et cette vérification comprend l’exécution des exemples; étant donné le très grand nombre de packages sur le CRAN, la vérification de chaque package séparément doit être rapide; - etc.
Pour rendre un package disponible sur Bioconductor, encore plus de lignes directrices à suivre s’ajoutent (voir https://www.bioconductor.org/developers/package-guidelines/).
Exemple :
Dans ces notes, nous allons reprendre l’exemple de la fonction dist_manhattan qui calcule la distance de Manhattan entre deux points provenant des notes sur les tests et exceptions en R. Modifions un peu la fonction dist_manhattan afin que les objets quelle retourne possèdent une classe. Ensuite, créons une fonction équivalente, nommée dist_manhattan_2, qui fait le même calcul, mais en appelant la fonction dist du package stats. L’objet retourné par cette fonction doit posséder la même classe qu’un objet retourné par dist_manhattan. Finalement, créons une méthode associée à cette classe pour la fonction générique print afin de formater l’impression d’un objet retourné par n’importe laquelle de nos deux fonctions. Voici le code source de ces fonctions, qui formeront le package que nous créerons.
dist_manhattan <- function(point1, point2) {
if (length(point1) != length(point2)) {
stop("'point1' and 'point2' must have the same length")
}
if (!is.null(dim(point1)) || !is.null(dim(point2))) {
warning("'point1' and 'point2' are treated as dimension 1 vectors")
}
sortie <- list(dist = sum(abs(point1 - point2)))
class(sortie) <- "distman"
return(sortie)
}dist_manhattan_2 <- function(point1, point2) {
call <- match.call()
dist <- stats::dist(rbind(point1, point2), method = "manhattan")
sortie <- list(dist = as.vector(dist), call = call)
class(sortie) <- "distman"
return(sortie)
}print.distman <- function(x, ...) {
cat("Manhattan distance between 'point1' and 'point2' :", x$dist, "\n", ...)
invisible(x)
}2 Étape 2. Créer l’arborescence de répertoires et fichiers du package
Un package, dans sa version source, est simplement une arborescence de répertoires et fichiers. Cependant, cette arborescence, ou structure, doit comprendre des fichiers et des sous-répertoires portant des noms et du contenu spécifiques.
Nous pouvons créer cette arborescence :
- manuellement (p. ex. dans un explorateur de fichiers offert par notre système d’exploitation),
- en appelant la fonction
package.skeletondu packageutils, - en appelant la fonction
create_packagedu packageusethis, chargé lors du chargement du packagedevtools(ce que nous ferons), - en utilisant les fonctionnalités de création de projets RStudio (ce qui résulte en fait en l’utilisation du package
usethis).
Peu importe la procédure utilisée, il faut d’abord avoir un répertoire principal portant le nom du package. Le contenu de base d’un répertoire de package est le suivant :
- sous-répertoire nommé
R: emplacement du code source R, - sous-répertoire nommé
man: emplacement du code source des fiches d’aide, - fichier nommé
DESCRIPTION: fichier de configuration, - fichier nommé
NAMESPACE: définition de l’espace de noms et de l’environnement des objets importés.
Nous pouvons représenter schématiquement ce contenu comme suit :
<nomPackage>/
|-- R/
|-- man/
|-- DESCRIPTION
|-- NAMESPACEPackage devtools
Concernant le package devtools, mentionnons qu’il comporte plusieurs fonctions facilitant la création et l’utilisation de packages R. Ce package est en quelque sorte sous-divisé en plus petits packages. Par exemple, les fonctions d’installation de package de devtools vues dans les notes sur l’utilisation de packages (dont le nom débute par install_) proviennent du package remotes. Les fonctions de configuration de packages, telle que create_package et d’autres fonctions que nous utiliserons plus loin, proviennent quant à elles du package usethis. Enfin, les fonctions permettant de lancer des commandes de vérification et de construction de packages proviennent directement du package devtools.
Pour la suite, le package devtools doit être installé sur notre ordinateur (ce qui implique que le package usethis y sera aussi installé). Nous ne le chargerons cependant pas. Nous allons plutôt appeler les fonctions distribuées par ce package en les précédant de l’opérateur :: et du nom de leur package exact de provenance, par exemple usethis::create_package().
2.1 Répertoire principal portant le nom du package
Premièrement, un nom doit être choisi pour le package. Ce nom peut contenir uniquement les caractères ASCII suivants : lettres, chiffres et . (point). Il ne peut donc pas contenir de lettres accentuées, ni le caractère _ (tiret bas ou underscore en anglais). Il doit être composé d’au moins 2 caractères, débuter par une lettre et ne pas terminer par un point. Ces règles sont tirées de la description du champ Package dans le fichier DESCRIPTION dans la documentation suivante : https://cran.r-project.org/doc/manuals/r-release/R-exts.html#The-DESCRIPTION-file
Notons que si nous souhaitons ultimement rendre notre package public sur le CRAN ou Bioconductor, il faut aussi s’assurer que le nom que nous avons choisi n’est pas déjà utilisé par un package sur cette plateforme. Le package R available permet de facilement vérifier la disponibilité d’un nom potentiel pour notre package.
Ensuite, utilisons la fonction usethis::create_package pour créer ce répertoire et sa structure de base. Le premier argument à fournir obligatoirement à la fonction usethis::create_package est l’emplacement désiré pour le répertoire sur notre ordinateur. Nous allons aussi demander à la fonction de créer un projet RStudio à partir du répertoire principal du package en donnant la valeur TRUE à l’argument rstudio. Appeler la fonction usethis::create_package à partir de RStudio a aussi cet impact. Le projet RStudio nous sera utile plus tard, lors de la construction du package. Finalement, nous utiliserons l’argument open = TRUE pour ouvrir le projet RStudio dans une nouvelle session R.
Exemple :
Dans notre exemple, choisissons le nom manhattan pour le package. Le répertoire principal du package sera un sous-répertoire de "C:/coursR/". Le chemin complet du répertoire principal du package, à fournir à la fonction create_package, est donc "C:/coursR/manhattan/".
usethis::create_package(path = "C:/coursR/manhattan/", rstudio = TRUE, open = TRUE)## √ Creating 'C:/coursR/manhattan/'
## √ Setting active project to 'C:/coursR/manhattan'
## √ Creating 'R/'
## √ Writing 'DESCRIPTION'
## Package: manhattan
## Title: What the Package Does (One Line, Title Case)
## Version: 0.0.0.9000
## Authors@R (parsed):
## * First Last <first.last@example.com> [aut, cre] (YOUR-ORCID-ID)
## Description: What the package does (one paragraph).
## License: `use_mit_license()`, `use_gpl3_license()` or friends to
## pick a license
## ## Encoding: UTF-8
## LazyData: true
## Roxygen: list(markdown = TRUE)
## RoxygenNote: 7.1.1
## √ Writing 'NAMESPACE'
## √ Writing 'manhattan.Rproj'
## √ Adding '^manhattan\\.Rproj$' to '.Rbuildignore'
## √ Adding '.Rproj.user' to '.gitignore'
## √ Adding '^\\.Rproj\\.user$' to '.Rbuildignore'
## √ Opening 'C:/coursR/manhattan/' in new RStudio session
## √ Setting active project to '<no active project>'Le travail réalisé par cette commande est bien décrit par l’impression produite. L’arborescence de répertoires et fichiers suivante a été produite :
manhattan/
|-- R/
|-- DESCRIPTION
|-- NAMESPACE
|-- manhattan.RprojLe répertoire créé contient aussi des fichiers et répertoires cachés, dont le nom débute par un point (notamment .Rproj.user/ et .Rbuildignore). Il s’agit de fichiers dont RStudio a besoin pour la gestion du projet, mais dont nous n’avons pas à nous soucier.
Toutes les manipulations futures décrites ci-dessous doivent être réalisées dans le projet RStudio de notre package en développement, qui vient d’être ouvert par l’appel à la fonction usethis::create_package.
2.2 Sous-répertoire R/
Le sous-répertoire R/ est l’endroit où placer les fichiers de scripts R (portant l’extension .R) contenant le code de création des fonctions. Ces fichiers doivent aussi contenir des commentaires roxygen si c’est l’outil utilisé pour générer la documentation, ce qui est le cas pour nous (nous y reviendrons plus loin).
Le développeur peut choisir les noms qu’il veut pour ces fichiers et il peut y répartir le code source comme il le veut. Évidemment, une bonne pratique est de répartir le code source de façon à ce que ce soit facile de s’y retrouver. Certains préconisent la stratégie « un fichier par fonction, portant le nom de la fonction », mais ce n’est pas obligatoire. La stratégie opposée, « un seul fichier contenant toutes les fonctions », représente rarement une bonne répartition du code, à moins que les fonctions soient très peu nombreuses.
Nous pouvons utiliser la fonction use_r du package usethis pour créer dans le sous-répertoire R des fichiers où nous enregistrerons le code source des fonctions. Cette fonction ouvre également le fichier.
Exemple :
Nous allons placer chacune des trois fonctions à mettre dans notre package dans trois fichiers séparés. Après cette étape, l’arborescence du package aura l’allure suivante :
manhattan/
|-- R/
|-- dist_manhattan.R
|-- dist_manhattan_2.R
|-- print.R
|-- DESCRIPTION
|-- NAMESPACE
|-- manhattan.RprojFichier dist_manhattan.R
usethis::use_r(name = "dist_manhattan")## √ Setting active project to 'C:/coursR/manhattan'
## * Modify 'R/dist_manhattan.R'
## * Call `use_test()` to create a matching test fileLe fichier dist_manhattan.R a été créé dans le sous-répertoire R. L’extension .R lui a été donnée par la fonction usethis::use_r. Il faut maintenant copier le code R suivant dans ce fichier et enregistrer le fichier.
dist_manhattan <- function(point1, point2) {
if (length(point1) != length(point2)) {
stop("'point1' and 'point2' must have the same length")
}
if (!is.null(dim(point1)) || !is.null(dim(point2))) {
warning("'point1' and 'point2' are treated as dimension 1 vectors")
}
sortie <- list(dist = sum(abs(point1 - point2)))
class(sortie) <- "distman"
return(sortie)
}Fichier dist_manhattan_2.R
usethis::use_r(name = "dist_manhattan_2")## * Modify 'R/dist_manhattan_2.R'
## * Call `use_test()` to create a matching test fileCopions le code R suivant dans le fichier ouvert et enregistrons le fichier.
dist_manhattan_2 <- function(point1, point2) {
call <- match.call()
dist <- stats::dist(rbind(point1, point2), method = "manhattan")
sortie <- list(dist = as.vector(dist), call = call)
class(sortie) <- "distman"
return(sortie)
}Fichier print.R
usethis::use_r(name = "print.distman")## * Modify 'R/print.R'
## * Call `use_test()` to create a matching test fileCopions le code R suivant dans le fichier R/print.R et enregistrons le fichier.
print.distman <- function(x, ...) {
cat("Manhattan distance between 'point1' and 'point2' :", x$dist, "\n", ...)
invisible(x)
}2.3 Sous-répertoire data/
Bien qu’il ne soit pas obligatoire, décrivons ici le sous-répertoire data/. Si le package doit contenir des données accessibles aux utilisateurs, c’est ici quelles doivent être placées. Elles doivent être stockées dans des objets R enregistrés dans des fichiers externes (un fichier par objet) sous un format propre à R (souvent .rda ou .RData). Ces données sont utiles, notamment, pour avoir des données à utiliser dans les exemples inclus dans les fiches d’aide.
La fonction use_data du package usethis facilite l’intégration de données à un package R. Illustrons comment l’utiliser dans l’exemple du package manhattan.
Notons qu’un package peut aussi contenir des données privées ou brutes (p. ex. un fichier texte comprenant des données). Nous ne verrons pas ici comment procéder pour intégrer ce type de données, mais voici une bonne source d’informations à ce sujet pour les intéressés : https://r-pkgs.org/data.html.
Exemple :
Créons un petit jeu de données aléatoire contenant 10 points en 2 dimensions.
points <- matrix(sample(1:10, size = 20, replace = TRUE), nrow = 10, ncol = 2)
colnames(points) <- c("X", "Y")Ajoutons cette matrice au package avec la commande suivante.
usethis::use_data(points)## √ Adding 'R' to Depends field in DESCRIPTION
## √ Creating 'data/'
## √ Saving 'points' to 'data/points.rda'
## * Document your data (see 'https://r-pkgs.org/data.html')L’arborescence de répertoires et fichiers du package est maintenant la suivante (toujours sans les répertoires et fichiers cachés) :
manhattan/
|-- R/
|-- dist_manhattan.R
|-- dist_manhattan_2.R
|-- print.R
|-- data/
|-- points.rda
|-- DESCRIPTION
|-- NAMESPACE
|-- manhattan.Rproj2.4 Sous-répertoire man/
Malgré le fait que celui-ci soit obligatoire, aucun sous-répertoire man/ n’a été inclus dans le répertoire principal du package par la fonction usethis::create_package. Ce sous-répertoire est celui destiné à contenir des fichiers source de fiches d’aide R (portant l’extension .Rd). Nous allons voir à la prochaine étape comment générer ces fichiers de façon automatique à l’aide du package roxygen2.
2.5 Fichier DESCRIPTION
Le fichier DESCRIPTION est très important pour la construction du package. C’est un fichier court, comportant de l’information de base décrivant l’utilité du package ainsi que ses configurations. Ce fichier doit respecter une syntaxe précise.
Par exemple, voici le gabarit de fichier DESCRIPTION qu’a produit l’appel à la fonction usethis::create_package (et qu’a modifié l’appel à la fonction usethis::use_data). Ce fichier doit être édité pour y insérer les bonnes informations.
Package: manhattan
Title: What the Package Does (One Line, Title Case)
Version: 0.0.0.9000
Authors@R:
person(given = "First",
family = "Last",
role = c("aut", "cre"),
email = "first.last@example.com",
comment = c(ORCID = "YOUR-ORCID-ID"))
Description: What the package does (one paragraph).
License: `use_mit_license()`, `use_gpl3_license()` or friends to
pick a license
Encoding: UTF-8
LazyData: true
Roxygen: list(markdown = TRUE)
RoxygenNote: 7.1.1
Depends:
R (>= 2.10)
Un fichier DESCRIPTION contient des champs nommés. Un champ débute par son nom (première lettre toujours majuscule), immédiatement suivi d’un deux-points et d’un espace. Vient ensuite la valeur fournie à ce champ. Les valeurs données aux champs peuvent s’étendre sur plus d’une ligne. De plus, l’ordre des champs n’est pas important.
Les champs obligatoires sont les suivants : Package, Version, License, Title, Description, Author, et Maintainer. Les champs Author, et Maintainer peuvent être remplacés par un seul champ Authors@R, comme dans le gabarit. Les codes de rôles acceptés dans ces champs sont énumérés dans la fiche d’aide ouverte par la commande help(person).
Voici quelques informations spécifiques à certains champs :
Package: le nom du package fourni dans ce champ doit correspondre parfaitement au nom du répertoire contenant les fichiers source du package;Version: il s’agit d’une séquence d’au minimum 2 (souvent 3, parfois même 4) nombres entiers non négatifs séparés par un seul caractère.(point) ou-(tiret);Maintainer: ce champ doit contenir un seul nom, celui de la personne à contacter pour toute question ou tout problème à rapporter concernant le package, suivi d’une adresse courriel valide placée entre les caractères<et>;Depends: ce champ est requis si le bon fonctionnement du package nécessite une certaine version minimale de R ou si d’autres packages doivent être chargés lors du chargement de notre package;Imports: ce champ contient la liste des autres packages à partir desquels des objets sont importés par le package (les packages d’où proviennent ceux-ci doivent être installés, mais ils n’ont pas besoin d’être chargés lors du chargement de notre package);LazyData: si la valeurtrueest fournie dans ce champ, les jeux de données contenus dans le package seront directement accessibles dans l’environnement du package, sans que l’utilisateur ait besoin d’appeler la fonctiondata(nous avions discuté des différentes façons d’accéder à des jeux de données dans un package dans les notes sur l’utilisation de packages R).
Le fichier DESCRIPTION doit contenir uniquement des caractères ASCII, sauf s’il contient un champ Encoding.
L’information complète et officielle concernant ce fichier peut être trouvée sur la page web suivante :
http://cran.r-project.org/doc/manuals/r-release/R-exts.html#The-DESCRIPTION-file
Le gabarit de fichier DESCRIPTION produit par la fonction usethis::create_package peut être modifié manuellement, en ouvrant le fichier (dans un éditeur de texte en RStudio par exemple), en remplaçant les informations génériques par les bonnes, puis en enregistrant le fichier. C’est la méthode que nous emploierons. Le package usethis possède aussi des fonctions permettant d’éditer le fichier DESCRIPTION par des commandes soumises dans la console, notamment :
- la fonction
use_description(pour modifier n’importe quel champ), - la fonction
use_package(pour modifier le champImportsou un autre champ relié aux dépendances à d’autres packages) et - des fonctions pour spécifier la licence utilisée (p. ex.
use_mit_license,use_gpl3_license, etc.) : ces fonctions ajoutent même un fichierLICENSEau répertoire principal du package.
Nous n’illustrerons pas ici l’utilisation de ces fonctions.
Exemple :
Voici le contenu du fichier DESCRIPTION à utiliser pour notre package en développement.
Package: manhattan
Title: Distance de Manhattan
Version: 1.0.0
Author: Sophie Baillargeon [aut, cre], Autre Auteur [aut]
Maintainer: Sophie Baillargeon <sophie.baillargeon@mat.ulaval.ca>
Description: Calcul de la distance de Manhattan entre deux points.
License: GPL-3
Encoding: UTF-8
LazyData: true
RoxygenNote: 7.1.1
Depends: R (>= 2.10)
Imports: stats
Le champ Depends est nécessaire uniquement en raison d’un détail technique. Nous avons inclus un jeu de données dans le package avec la fonction usethis::use_data. Celle-ci a en fait utilisé la fonction save pour créer le fichier .rda contenant les données. Cependant, le format de fichier créé par cette fonction a légèrement changé au fil des ans. La fonction usethis::use_data a par défaut utilisé un format qui peut seulement être compris par une version de R égale ou plus récente que 2.10. Il s’agit d’une très vieille version de R. Ce n’est donc pas vraiment une contrainte de dépendre de l’utilisation d’une version de R >= 2.10.
Le champ Imports, contenant ici le package stats, est requis puisque la fonction dist_manhattan_2 utilise une fonction de ce package.
Notons que le champ Authors@R suivant pourrait remplacer les champs Author et Maintainer précédents.
Authors@R: c(
person(given = "Sophie", family = "Baillargeon", role = c("aut", "cre"),
email = "sophie.baillargeon@mat.ulaval.ca"),
person(given = "Autre", family = "Auteur", role = "aut"))2.6 Fichier NAMESPACE
Le fichier NAMESPACE permet de définir quels objets sont accessibles dans un package, c’est-à-dire exportés de l’espace de noms du package. Ce fichier permet aussi d’identifier quels objets provenant d’autres packages sont utilisés dans le package, donc de définir le contenu de l’environnement des objets importés par le package. Nous allons voir comment utiliser roxygen2 pour générer de façon automatique ce fichier.
2.7 Autres fichiers et répertoires
Un package peut contenir plusieurs autres fichiers et sous-répertoires, comme nous avons pu le constater avec le package ggplot2. Voici quelques autres répertoires parfois nécessaires :
- Sous-répertoire nommé
src: Si le package contient du code C, C++ ou Fortran, ce code doit se trouver dans ce répertoire. - Sous-répertoire nommé
vignettes: Si le package contient de la documentation autre que les fiches d’aide (par exemple un guide d’utilisateur), il devrait idéalement se trouver dans ce répertoire. - Sous-répertoire nommé
tests: Des tests, par exemple écrits avectestthat, peuvent être intégrés au package dans ce sous-répertoire et exécutés à chaque fois que le package est vérifié (checkdécrit à l’étape 4). Pour en savoir plus : https://r-pkgs.org/tests.html#test-structure - etc. : http://cran.r-project.org/doc/manuals/r-release/R-exts.html#Package-subdirectories
Autres fichiers utiles :
- Fichier nommé
NEWSouNEWS.md: Ce fichier décrit les modifications apportées à un package lors d’une mise à jour. Plusieurs types de mise en forme sont acceptés pour ce fichier. - etc. : http://cran.r-project.org/doc/manuals/r-release/R-exts.html#Package-structure
3 Étape 3. Écrire la documentation des fonctions et du package
Documenter ses fonctions est une étape intégrée au développement des fonctions. Cependant, il faut maintenant écrire la documentation dans un format qui produira correctement les fiches d’aide qui doivent être incluses dans le package.
Ces fiches d’aide proviennent en fait de fichiers portant l’extension .Rd. Cependant, nous ne verrons pas comment éditer directement ces fichiers. Nous allons plutôt apprendre à utiliser le package roxygen2, qui permet de générer des fichiers .Rd, ainsi que le fichier NAMESAPCE, de façon automatique, à partir de commentaires intégrés au code.
L’utilisation de roxygen2 comporte les avantages suivants :
- la documentation se situe dans le même fichier que le code, ce qui aide à se rappeler que la documentation doit être mise à jour si le code est modifié,
- la syntaxe
roxygen2est un peu plus simple que la syntaxe des fichiers.Rd(qui sera illustrée plus loin).
Rappelons que dans un package il faut obligatoirement documenter :
Les fonctions publiques
Il est essentiel de faire des fiches d’aide pour ces fonctions, afin que tout utilisateur comprenne comment appeler correctement la fonction. Si un utilisateur a mal compris la documentation et fournit par erreur une valeur d’argument invalide en entrée, il est aussi souhaitable que la fonction retourne une erreur informative. Le code de ces fonctions publiques comporte donc typiquement de la validation des valeurs fournies en entrée aux arguments.
À l’opposé, les fonctions privées ou internes ne sont pas conçues pour être appelées par n’importe quel utilisateur. Ces fonctions ne sont pas exportées de l’espace de noms et elles ne sont pas documentées officiellement. Il est tout de même bon de documenter minimalement ces fonctions pour nous-mêmes, mais nous n’avons pas à produire de fiches d’aide pour elles. De plus, pour ne pas alourdir ces fonctions, elles comportent typiquement peu ou pas de validation d’arguments.
Les jeux de données dans le répertoire data/
Chaque jeu de données dans le répertoire data d’un package doit être décrit dans une fiche d’aide pour expliquer son contenu.
Les classes et méthodes S4 ou RC publiques
Les packages exploitant le système de programmation orientée objet S4 ou le système RC doivent fournir des fiches d’aide pour les classes et méthodes publiques. Nous ne verrons pas ici comment écrire cette documentation, car nous utiliserons seulement le système orienté objet S3. Plus d’information peut être trouvée sur les pages web suivantes :
Il est aussi recommandé de documenter :
Les méthodes S3 pour des fonctions génériques :
Nous pouvons leur faire une fiche d’aide indépendante ou encore les documenter dans la même fiche d’aide que la fonction qui crée les objets de la classe en question.Le package lui-même :
Il est utile de créer une fiche d’aide présentant le package.
3.1 Documentation avec roxygen2
Comment écrire de la documentation avec roxygen2 ? Il suffit d’insérer des « commentaires » roxygen dans le code source des fonctions, donc dans les scripts situés dans le sous-répertoire R. Un commentaire roxygen débute par #', ce qui le distingue d’un commentaire ordinaire, qui débute par #. Il contient des tags ayant des significations particulières.
RStudio possède une fonctionnalité permettant d’ajouter un gabarit de commentaires roxygen en entête de nos fonctions. Cette fonctionnalité est accessible par le menu « Code > Insert Roxygen Skeleton ».
Exemple :
D’abord, voici ce que nous obtenons si nous demandons à RStudio d’insérer un gabarit de commentaires roxygen en entête de la fonction dist_manhattan.
Fichier C:/coursR/manhattan/R/dist_manhattan.R :
#' Title
#'
#' @param point1
#' @param point2
#'
#' @return
#' @export
#'
#' @examples
dist_manhattan <- function(point1, point2) {
if (length(point1) != length(point2)) {
stop("'point1' and 'point2' must have the same length")
}
if (!is.null(dim(point1)) || !is.null(dim(point2))) {
warning("'point1' and 'point2' are treated as dimension 1 vectors")
}
sortie <- list(dist = sum(abs(point1 - point2)))
class(sortie) <- "distman"
return(sortie)
}
Après adaptation, voici de quoi ce fichier pourrait avoir l’air.
Fichier C:/coursR/manhattan/R/dist_manhattan.R :
#' Distance de Manhattan
#'
#' Calcule la distance de Manhattan entre deux points
#'
#' @param point1 Un vecteur numerique des coordonnees du premier point.
#' @param point2 Un vecteur numerique des coordonnees du deuxieme point.
#'
#' @return une seule valeur : la distance de Manhattan entre
#' \code{point1} et \code{point2}
#' @author Sophie Baillargeon
#' @export
#' @examples
#' dist_manhattan(point1 = points[4, ], point2 = points[8, ])
dist_manhattan <- function(point1, point2) {
if (length(point1) != length(point2))
stop("'point1' and 'point2' must have the same length")
if (!is.null(dim(point1)) || !is.null(dim(point2)))
warning("'point1' and 'point2' are treated as dimension 1 vectors")
sortie <- list(dist = sum(abs(point1 - point2)))
class(sortie) <- "distman"
return(sortie)
}
Des explications concernant les différents tags se retrouvent après les exemples. Avant voyons aussi les commentaires roxygen que nous pourrions utiliser pour les fonctions dist_manhattan_2 et print.distman.
Fichier C:/coursR/manhattan/R/dist_manhattan_2.R :
#' Distance de Manhattan
#'
#' Calcule la distance de Manhattan entre deux points
#'
#' Utilise la fonction \code{\link[stats]{dist}} du package \pkg{stats}.
#'
#' @param point1 Un vecteur numerique des coordonnees du premier point.
#' @param point2 Un vecteur numerique des coordonnees du deuxieme point.
#'
#' @return \item{dist}{ la distance de Manhattan entre \code{point1} et \code{point2} }
#' @return \item{call}{ une copie de l'appel de la fonction }
#' @author Sophie Baillargeon
#' @export
#' @importFrom stats dist
#' @examples
#' dist_manhattan_2(point1 = c(0,-5, 3), point2 = c(2,-15, 4))
dist_manhattan_2 <- function(point1, point2) {
call <- match.call()
dist <- stats::dist(rbind(point1, point2), method = "manhattan")
sortie <- list(dist = as.vector(dist), call = call)
class(sortie) <- "distman"
return(sortie)
}
Fichier C:/coursR/manhattan/R/print.R :
#' @describeIn dist_manhattan Affiche un objet de classe \code{"distman"}
#' @param x Un objet produit par la fonction \code{dist_manhattan} ou
#' \code{dist_manhattan_2}, a afficher.
#' @param \dots D'autres arguments passes a d'autres methodes.
#' @export
print.distman <- function(x, ...) {
cat("Manhattan distance between 'point1' and 'point2' :", x$dist, "\n", ...)
invisible(x)
}
Finalement, pour produire une fiche d’aide minimalise pour le package lui-même, nous pourrions utiliser la fonction use_package_doc du package usethis.
usethis::use_package_doc()## √ Writing 'R/manhattan-package.R'
## * Modify 'R/manhattan-package.R'Cette commande à créer le fichier manhattan-package.R dans le répertoire R. Voici un aperçu de son contenu initial.
#' @keywords internal
"_PACKAGE"
# The following block is used by usethis to automatically manage
# roxygen namespace tags. Modify with care!
## usethis namespace: start
## usethis namespace: end
NULLNous ne retoucherons pas ici à ces commentaires. La fiche d’aide du package contiendra uniquement une copie des champs Title et Description du fichier DESCRIPTION. Nous pourrions évidemment y ajouter plus d’informations.
Il faut ajouter quelque part un dernier bloc de commentaires roxygen pour documenter le jeu de données inclus dans le package. Cet ajout pourrait être fait dans n’importe quel fichier du répertoire R. Nous ferons l’ajout dans le fichier manhattan-package.R.
Fichier C:/coursR/manhattan/R/manhattan-package.R après l’ajout :
#' @keywords internal
"_PACKAGE"
# The following block is used by usethis to automatically manage
# roxygen namespace tags. Modify with care!
## usethis namespace: start
## usethis namespace: end
NULL
#' Points aleatoires
#'
#' Coordonnes en deux dimensions de 10 points aleatoires.
#'
#' @format Une matrice contenant 10 points designes par les
#' coordonnees suivantes.
#' \describe{
#' \item{\code{X}}{ coordonnee en X }
#' \item{\code{Y}}{ coordonnee en Y }
#' }
#' @examples
#' dist_manhattan(point1 = points[4, ], point2 = points[8, ])
#'
#' # Note : Ces donnees ont ete crees par les instructions suivantes
#' points <- matrix(sample(1:10, size = 20, replace = TRUE),
#' nrow = 10, ncol = 2)
#' colnames(points) <- c("X", "Y")
"points"
Explications :
Pour les fonctions et les méthodes associées à des fonctions génériques, il suffit de mettre en entête du code source les commentaires roxygen qui généreront la documentation. Pour le package et les jeux de données, il est recommandé d’ajouter un ou des fichiers d’extension .R dans le répertoire R. Ces fichiers doivent contenir les commentaires roxygen pour documenter globalement le package, suivi de l’instruction "_PACKAGE", et les commentaires pour documenter les jeux de données, chaque bloc suivi du nom du jeu de données sous forme de chaîne de caractères (par exemple "points") .
La première phrase d’un bloc de commentaires roxygen, si elle n’est précédée d’aucun tag roxygen, sera interprétée comme le titre de la fiche d’aide. Ce titre peut aussi être précédé du tag @title.
Si le titre est suivi d’une ligne de commentaire roxygen vide, puis de lignes de texte non précédées d’un tag, celles-ci constitueront la section Description. Ce paragraphe peut aussi être précédé du tag @description.
Si le paragraphe est aussi suivi d’une ligne de commentaire roxygen vide, tout le texte après cette ligne vide, mais avant les lignes débutant par un tag roxygen formera la section Details. Ces paragraphes peuvent optionnellement être précédés du tag @details.
Ainsi, les deux documentations suivantes sont équivalentes :
#' Distance de Manhattan
#'
#' Calcule la distance de Manhattan entre deux points
#'
#' Utilise la fonction \code{\link[stats]{dist}} du package \pkg{stats}.
#'
#' @param point1 Un vecteur numerique des coordonnees du premier point.
#' @param point2 Un vecteur numerique des coordonnees du deuxieme point.
#' @return \item{dist}{ la distance de Manhattan entre \code{point1} et \code{point2} }
#' @return \item{call}{ une copie de l'appel de la fonction }
#' @author Sophie Baillargeon
#' @export
#' @importFrom stats dist
#' @examples
#' dist_manhattan_2(point1 = c(0,-5, 3), point2 = c(2,-15, 4))
dist_manhattan_2 <- function(point1, point2) {
# ... (code omis ici)
}et
#' @title Distance de Manhattan
#' @description Calcule la distance de Manhattan entre deux points
#' @details Utilise la fonction \code{\link[stats]{dist}} du package \pkg{stats}.
#' @param point1 Un vecteur numerique des coordonnees du premier point.
#' @param point2 Un vecteur numerique des coordonnees du deuxieme point.
#' @return \item{dist}{ la distance de Manhattan entre \code{point1} et \code{point2} }
#' @return \item{call}{ une copie de l'appel de la fonction }
#' @author Sophie Baillargeon
#' @export
#' @importFrom stats dist
#' @examples
#' dist_manhattan_2(point1 = c(0,-5, 3), point2 = c(2,-15, 4))
dist_manhattan_2 <- function(point1, point2) {
# ... (code omis ici)
}Les sections qui suivent le titre, la description (ces sections sont requises) et les informations détaillées (cette section est optionnelle), doivent toutes débuter par des tags. Il faut obligatoirement décrire, si la fonction en possède :
- les arguments en entrée avec le tag
@param:
il faut une description par argument, de la forme@param <nom_argument> <description>, où la description peut s’étendre sur plusieurs lignes; - la sortie avec le tag
@return:
si la fonction retourne une liste, il est recommandé d’avoir une description par élément de la liste, de la forme@return \item{<nom_element_liste>}{<description>}, où la description peut encore une fois s’étendre sur plusieurs lignes.
Il est recommandé de toujours mettre un ou des exemples dans la fiche d’aide d’une fonction ou d’un jeu de données. Dans la documentation roxygen, ceux-ci doivent être ajoutés à la fin du bloc de documentation, après le tag @examples.
Nous pouvons aussi ajouter les informations suivantes :
- les noms des auteurs avec le tag
@author, - des références avec le tag
@references, - des liens vers les fiches d’aide de fonction en lien avec la fonction documentée avec le tag
@seealso, - etc. voir https://roxygen2.r-lib.org/articles/rd.html
Documenter plusieurs fonctions dans la même fiche
Le tag @describeIn sert à présenter plusieurs fonctions dans la même fiche d’aide. Dans l’exemple, la fonction dist_manhattan et la méthode print.distman sont documentées dans la même fiche d’aide grâce au tag @describeIn. Ce tag doit être suivi du nom de la fiche dans laquelle de l’information doit être ajoutée, puis d’une courte description à propos de la fonction ou méthode documentée.
Le tag @rdname permet également de regrouper des fiches d’aide (voir https://roxygen2.r-lib.org/articles/rd.html#documenting-multiple-functions-in-the-same-file-1 pour plus d’information).
Documenter des jeux de données
Pour documenter des jeux de données, deux tags supplémentaires sont disponibles :
@formatpour décrire la structure R qui contient les données,@sourcepour fournir une référence concernant la provenance des données.
Nom des fiches d’aide
Nous pouvons contrôler le nom des fiches d’aide avec le tag @name. Sans ce tag, la fiche d’aide porte le nom de la fonction ou le nom du jeu de données qui suit le bloc de documentation roxygen. Pour une documentation de package, l’instruction "_PACKAGE" qui suit le bloc de documentation indique à roxygen2 d’utiliser le nom du package suivi de -package comme nom de fiche d’aide. Dans l’exemple, le nom de la fiche d’aide du package est donc manhattan-package. Cependant, roxygen2 crée aussi un alias pour le nom de la fiche d’aide qui est le nom du package.
Pour ouvrir une fiche d’aide avec la fonction help ou l’opérateur ?, il faut lui donner en entrée le nom de la fiche ou un alias du nom de la fiche. Il est donc important de nommer intelligemment nos fiches d’aide. La norme est de donner à une fiche d’aide le nom de l’objet principal qu’elle documente et d’ajouter des alias pour les noms des autres objets documentés dans la fiche (c’est ce que fait roxygen2 de façon automatique lorsque les tags @describeIn et @rdname sont utilisés).
Texte formaté dans des fiches d’aide
Dans les commentaires roxygen, il est possible de formater du texte en utilisant les tags de mises en forme acceptés dans les fichiers .Rd. Cette possibilité été exploitée dans les exemples précédents. Ces tags, par exemple \code{}, \pkg{}, \item{}, \link{}, \dots, etc., sont documentés sur la page web suivante : https://cran.r-project.org/doc/manuals/r-release/R-exts.html#Marking-text
Depuis la version 6.0.0 de roxygen2, il est également possible d’utiliser la syntaxe Markdown dans des commentaires roxygen pour formater le texte. La fonction usethis::create_package avait d’ailleurs considéré par défaut que nous utiliserions cette option en ajoutant le champ Roxygen: list(markdown = TRUE) dans le fichier DESCRIPTION. Nous avons cependant retiré ce champ. Plus d’information à propos de l’utilisation de la syntaxe Markdown dans des commentaires roxygen peut être trouvée ici : https://roxygen2.r-lib.org/articles/rd-formatting.html
3.2 Générer les fichiers .Rd et le fichier NAMESPACE
Pour générer les fichiers .Rd et le fichier NAMESPACE, il suffit d’appeler la fonction roxygenize du package roxygen2. La fonction document du package devtools ou des fonctionnalités de RStudio peuvent aussi appeler cette fonction pour nous.
Exemple :
devtools::document()## Updating manhattan documentation
## First time using roxygen2. Upgrading automatically...
## Loading manhattan
## Writing NAMESPACE
## Writing NAMESPACE
## Writing dist_manhattan.Rd
## Writing dist_manhattan_2.Rd
## Writing manhattan-package.Rd
## Writing points.Rd
Dans l’exemple présenté précédemment, les commentaires roxygen des fichiers dist_manhattan.R, dist_manhattan_2.R, print.R et manhattan-package.R, du sous-répertoire C:/coursR/manhattan/R, ont produit (ou modifié) les fichiers suivants :
NAMESPACEdans le répertoireC:/coursR/manhattan/;dist_manhattan.Rd,dist_manhattan_2.Rd,points.Rdetmanhattan-package.Rddans le répertoireC:/coursR/manhattan/man/.
L’arborescence de répertoires et fichiers du package est donc maintenant la suivante.
manhattan/
|-- R/
|-- dist_manhattan.R
|-- dist_manhattan_2.R
|-- print.R
|-- manhattan-package.R
|-- man/
|-- dist_manhattan.Rd
|-- dist_manhattan_2.Rd
|-- manhattan-package.Rd
|-- points.Rd
|-- data/
|-- points.rda
|-- DESCRIPTION
|-- NAMESPACE
|-- manhattan.Rproj
Le fichier C:/coursR/manhattan/NAMESPACE est maintenant le suivant :
# Generated by roxygen2: do not edit by hand
S3method(print,distman)
export(dist_manhattan)
export(dist_manhattan_2)
importFrom(stats,dist)
Voici de quoi a l’air un des fichiers d’extension .Rd généré.
Fichier "C:/coursR/manhattan/man/dist_manhattan.Rd" :
% Generated by roxygen2: do not edit by hand
% Please edit documentation in R/dist_manhattan.R, R/print.R
\name{dist_manhattan}
\alias{dist_manhattan}
\alias{print.distman}
\title{Distance de Manhattan}
\usage{
dist_manhattan(point1, point2)
\method{print}{distman}(x, ...)
}
\arguments{
\item{point1}{Un vecteur numerique des coordonnees du premier point.}
\item{point2}{Un vecteur numerique des coordonnees du deuxieme point.}
\item{x}{Un objet produit par la fonction \code{dist_manhattan} ou
\code{dist_manhattan_2}, a afficher.}
\item{\dots}{D'autres arguments passes a d'autres methodes.}
}
\value{
une seule valeur : la distance de Manhattan entre
\code{point1} et \code{point2}
}
\description{
Calcule la distance de Manhattan entre deux points
}
\section{Methods (by generic)}{
\itemize{
\item \code{print}: Affiche un objet de classe \code{"distman"}
}}
\examples{
dist_manhattan(point1 = points[4, ], point2 = points[8, ])
}
\author{
Sophie Baillargeon
}
Ces fichiers utilisent une syntaxe inspirée de la syntaxe de LaTeX. Lorsque nous utilisons roxygen2, nous n’avons pas besoin d’éditer directement ces fichiers, donc nous n’avons pas besoin de comprendre leur syntaxe (à l’exception peut-être des tags de mise en forme). La syntaxe roxygen est plus simple.
Après construction et installation du package, cette fiche d’aide aura l’allure suivante :
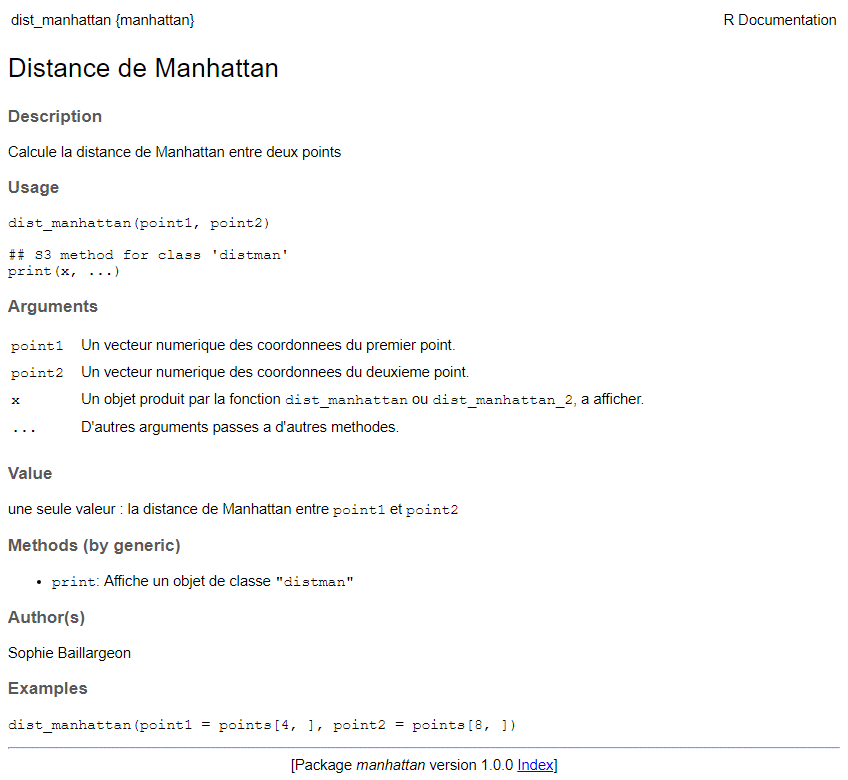
4 Étape 4. Vérifier et construire le package
Une fois les étapes 1 à 3 complétées, nous sommes prêts à construire le fichier compressé du package à partir du répertoire contenant les fichiers source. En fait, nous allons d’abord à cette étape vérifier le répertoire afin de nous assurer qu’il produira un package conforme.
Ces tâches se réalisent avec les utilitaires R :
R CMD check,R CMD build,R CMD INSTALL.
Ces utilitaires s’exploitent par des commandes soumises dans le terminal sous Linux ou macOS et dans une fenêtre invite de commandes sous Windows. Dans ces notes, nous verrons comment utiliser un outil qui soumet ces commandes à notre place, sans que nous ayons besoin d’ouvrir le terminal ou la fenêtre d’invite de commandes.
Les utilitaires R CMD check, build et INSTALL sont inclus dans l’installation de base de R (dans le package utils). Cependant, ils nécessitent des outils supplémentaires pour fonctionner : les outils de développement de logiciel GNU, incluant un compilateur C/C++. Voici comment s’assurer que ces outils sont installés sur notre ordinateur, selon le système d’exploitation (plus de détails sur la page https://support.rstudio.com/hc/en-us/articles/200486498-Package-Development-Prerequisites)\ :
Windows : il faut installer les « Rtools », téléchargeable sur la page web https://cran.r-project.org/bin/windows/Rtools/ (plus de détails dans le Guide d’installation ou de mise à jour de R et RStudio);
macOS : il faut installer les « Xcode command line tools », disponibles gratuitement, s’ils ne sont pas déjà installés (plus de détails dans le Guide d’installation ou de mise à jour de R et RStudio);
Linux : il faut s’assurer d’avoir installé R accompagné des ses « development tools » (
r-base-dev).
Le package pkgbuild comporte une fonction pour tester si tout le nécessaire au développement de package est installé et fonctionne correctement.
pkgbuild::has_build_tools()Si cet appel à la fonction pkgbuild::has_build_tools retourne TRUE, alors tout est fonctionnel.
De plus, une des commandes R pour le développement de packages, soit R CMD check, a besoin d’une installation de LaTeX pour tester la création de la documentation des packages en format PDF. Pour développer des packages, vous avez donc le choix entre :
- installer LaTeX sur votre ordinateur (tel que mentionné dans le Guide d’installation ou de mise à jour de R et RStudio) :
- un compilateur LaTeX multiplateforme gratuit est MiKTeX (https://miktex.org/download),
- un autre compilateur LaTeX gratuit pour macOS est MacTeX (http://www.tug.org/mactex/),
- les systèmes Linux viennent habituellement par défaut avec une distribution de LaTeX;
- omettre la création de la documentation PDF lors de la soumission de la commande
R CMD checkgrâce à l’option--no-manual(plus d’informations à venir en temps opportun).
4.1 Lancement des commandes de vérification et de construction avec devtools
Les commandes de construction et de vérification d’un package peuvent être soumises à l’aide de fonctions du package devtools. Ces fonctions permettent de soumettre des commandes R CMD sans passer par le terminal ou l’invite de commandes, ce qui simplifie vraiment le processus. Avec devtools, tout se réalise via des commandes soumises dans la console R.
4.1.1 Vérification de package avec la fonction devtools::check
Il est recommandé de d’abord vérifier le package avant de le construire. Cette vérification se lance avec devtools grâce à la fonction check.
4.1.1.1 Exemple :
devtools::check()L’impression produite est plutôt longue et n’est donc pas présentée ici. Si des erreurs sont détectées, il faut les régler. Les messages d’erreur peuvent nous aider à comprendre le problème. Faire une recherche internet avec le message d’erreur intégral (p. ex. encadré de guillemets dans Google) est aussi souvent informatif.
Nous souhaitons aussi éviter les avertissements. Il est recommandé de les régler autant que possible.
Par défaut, la fonction devtools::check appelle d’abord la fonction devtools::document, ce qui assure que la documentation est à jour avant de vérifier le package.
Notons aussi que la fonction devtools::check possède un argument cran prenant la valeur par défaut TRUE, ce qui signifie que la commande R CMD check est lancée avec l’option --as-cran. Pour omettre cette option, il faut appeler la fonction devtools::check comme suit :
devtools::check(cran = FALSE)Note : Si vous n’avez pas de compilateur LaTeX sur votre ordinateur, ajoutez aussi l’argument manual = FALSE. Cette fonction indique à R CMD check de ne pas vérifier la compilation de la version PDF de la documentation du package.
4.1.2 Construction de package avec la fonction devtools::build
Une fois satisfaits des résultats de la vérification, nous pouvons passer à l’étape ultime : construire le package. Cette étape permet d’obtenir une archive (fichier compressé) contenant le package. Nous avons vu dans les notes sur l’utilisation de packages R que ceux-ci sont distribués sous trois formats d’archive différents :
.tar.gz(package source) pour Linux,.zippour Windows,.tgzpour macOS.
Tous ces formats d’archive peuvent être créés avec la fonction build du package devtools. Pour créer le package source (.tar.gz), aucun argument n’a à être fourni en entrée à la fonction. Celle-ci lance en fait la commande R CMD build et un fichier nommé "nomPackage_numeroVersion.tar.gz" (dans notre exemple
manhattan_1.0.0.tar.gz) est créé dans le répertoire contenant le répertoire principal du package (donc le répertoire parent du répertoire portant le nom du package).
Les archives de format .zip et .tgz sont des versions dites binaires d’un package. La fonction devtools::build avec l’argument binary = TRUE permet de les créer. Avec cet argument, cette fonction lance en fait la commande R CMD INSTALL --build. Si cette commande est lancée à partir d’un système d’exploitation Windows, une archive .zip est créée, alors que c’est plutôt une archive .tgz qui est créée lorsque la commande est lancée à partir d’un système d’exploitation macOS. Le fichier sera nommé nomPackage_numeroVersion.zip (sous Windows) ou nomPackage_numeroVersion.tgz (sous macOS) et créé dans le répertoire au-dessus du répertoire principal du package.
Exemple :
Voici ce que j’obtiens en créant les versions source et binaire de notre package sur mon ordinateur, qui est sous système d’exploitation Windows.
devtools::build()## √ checking for file 'C:\coursR\manhattan/DESCRIPTION' (435ms)
## - preparing 'manhattan': (374ms)
## √ checking DESCRIPTION meta-information ...
## - checking for LF line-endings in source and make files and shell scripts
## - checking for empty or unneeded directories
## - building 'manhattan_1.0.0.tar.gz'
##
## [1] "C:/coursR/manhattan_1.0.0.tar.gz"devtools::build(binary = TRUE)## - installing to library 'C:/Users/Sophie/AppData/Local/Temp/Rtmpwpubsf/temp_libpath3d403f3b5c1' (375ms)
## - installing *source* package 'manhattan' ...
## ** using staged installation
## ** R
## ** data
## *** moving datasets to lazyload DB
## ** byte-compile and prepare package for lazy loading
## ** help
## *** installing help indices
## converting help for package 'manhattan'
## finding HTML links ... done
## dist_manhattan html
## dist_manhattan_2 html
## manhattan-package html
## points html
## ** building package indices
## ** testing if installed package can be loaded from temporary location
## *** arch - i386
## *** arch - x64
## ** testing if installed package can be loaded from final location
## *** arch - i386
## *** arch - x64
## ** testing if installed package keeps a record of temporary installation path
## - MD5 sums
## packaged installation of 'manhattan' as manhattan_1.0.0.zip
## - DONE (manhattan)
## [1] "C:/coursR/manhattan_1.0.0.zip"4.1.3 Accès à nos fonctions en cours de développement
Pour charger la définition de nos fonctions en cours de développement, par exemple pour les tester interactivement, la fonction load_all du package devtools est utile. Elle simule un chargement du package avec library sans réellement installer le package.
Pour vraiment installer le package, nous pouvons utiliser la fonction install du package devtools. La feuille de triche du package devtools décrit schématiquement les différences entre ces fonctions : https://rawgit.com/rstudio/cheatsheets/master/package-development.pdf
4.2 Lancement des commandes de vérification et de construction en RStudio
RStudio peut aussi lancer pour nous les commandes de construction et de vérification de packages.
Cette sous-section explique comment réaliser avec des menus de RStudio ce que nous venons de réaliser avec des fonctions du package devtools. Il s’agit d’une méthode de remplacement en cas de problèmes. Si tout a bien fonctionné à la sous-section précédente, ce qui est présenté ici ne nous est pas utile.
Sous-étape a) Créer un projet RStudio avec le répertoire principal du package
Si nous n’avions pas utilisé la fonction usethis::create_package pour créer le répertoire principal du package, celui-ci ne serait peut-être pas un projet RStudio. S’il ne l’était pas, il faudrait tout d’abord créer un projet RStudio avec le répertoire principal de notre package. Pour ce faire, nous pourrions procéder comme suit :
- ouvrir le menu « File » et sélectionner « New Project… » (il y a aussi un bouton dans la barre de RStudio en haut à droite, nommé à l’origine « Project: (None) » qui ouvre un menu contenant aussi l’élément « New Project… »),
- sélectionner « Existing Directory »,
- sélectionner le répertoire principal du package, c’est-à-dire le répertoire portant le nom du package et contenant les fichiers source,
- cliquer sur « Create Project ».
Le projet sera créé et ouvert. Ça ajoute des fichiers dans le répertoire de notre package. Nous ne nous préoccuperons pas de ces fichiers. Ils sont automatiquement ignorés lors de la construction du package avec RStudio.
Note : Si nous sélectionnons « New Directory » plutôt que « Existing Directory », puis « R package », RStudio crée un squelette de répertoire de fichiers source de package.
Sous-étape b) Configurer les options de RStudio
Voici quelques configurations de RStudio que je conseille d’utiliser.
Options globales :
(à modifier une seule fois)
- par le menu « Tools > Global Options… »,
- dans « Packages », décocher « Cleanup output after successful R CMD check ».
Les répertoires générés par la commande R CMD check ne seront ainsi pas effacés et nous pourrons, par exemple, aller y récupérer la version PDF de la documentation du package.
Options du projet :
(à modifier pour chaque nouveau projet)
- par le menu « Tools > Project Options… »;
- dans « Build Tools » :
- cocher « Generate documentation with Roxygen »,
- si le menu de configuration ne s’ouvre pas automatiquement, cliquez sur « Configure… », puis assurez-vous que les options suivantes soient cochées :
- « Use roxygen to generate » : « Rd files » et « NAMESPACE »,
- « Automatically roxygenize when running » : tout cocher.
Avec ces dernières configurations, la majorité des commandes de construction et de vérification de package lancées par le menu « Build » (voir ci-dessous) vont d’abord soumettre la commande roxygenize sur le répertoire du package avant de faire leur travail. Ainsi, les fichiers .Rd de documentation et le fichier NAMESPACE seront mis à jour à chaque lancement d’une de ces commandes
Nous pouvons aussi soumettre la commande roxygenize par le menu « Build > Document ».
Note : Si vous n’avez pas de compilateur LaTeX sur votre ordinateur, ajoutez l’option suivante à R CMD check : --no-manual. Cette fonction indique à R CMD check de ne pas vérifier la compilation de la version PDF de la documentation du package.
5 Étape 5. Si désiré, partager le package
Notre package est maintenant prêt à être utilisé. Si nous souhaitons le partager avec le grand public, nous pouvons le rendre disponible sur le CRAN. Pour ce faire, il faut d’abord s’assurer de respecter les politiques du CRAN : http://cran.r-project.org/web/packages/policies.html. Comme mentionné précédemment, il faut donc que notre package passe le R CMD check --as-cran sans erreurs ni avertissements.
Une fois s’être assuré de respecter les politiques du CRAN, nous pouvons soumettre notre package au CRAN en ligne par l’intermédiaire de l’interface web suivante : https://cran.r-project.org/submit.html
Il suffit de suivre les instructions.
6 Étape 6. Au besoin, mettre à jour le package
Mettre à jour un package signifie de le modifier pour ajouter des fonctionnalités et/ou corriger des bogues. Lors d’une mise à jour, il faut suivre les étapes suivantes.
Sous-étape a) Incrémenter le numéro de version :
Cette incrémentation doit être effectuée dans le fichier DESCRIPTION, ainsi que dans tout commentaire roxygen mentionnant la date de construction du package, par exemple dans la fiche d’aide du package.
Rappelons qu’un numéro de version est une séquence d’au minimum 2 (souvent 3, parfois 4) nombres entiers non négatifs séparés par un seul caractère caractère . (point) ou - (tiret). Ces nombres ne sont pas contraints à être compris entre 0 et 9.
Voici les règles que plusieurs développeurs de packages R suivent dans la numérotation des versions de leurs packages. Ils forment les numéros de version de 3 nombres entiers séparés par un point. En cours de développement, soit avant d’avoir une version considérée suffisamment testée, ils utilisent un 0 comme premier nombre dans le numéro de version (par exemple 0.9.12). Lorsqu’ils jugent leur package assez fiable pour être rendu disponible à plus grande échelle qu’à l’interne, ils changent le premier nombre dans le numéro de version pour 1, ce qui fait retomber à zéro les nombres suivants. La première version officielle porte donc le numéro de version 1.0.0.
Ensuite, ils font évoluer les numéros de version comme suit.
Lors d’une mise à jour majeure (beaucoup de nouvelles fonctionnalités) : le premier nombre de la numérotation est incrémenté de 1, les nombres subséquents retombent à 0.
Lors d’une mise à jour mineure (seulement quelques fonctionnalités pas trop importantes ont été ajoutées ou d’importants bogues ont été réglés) : le premier nombre est inchangé, par contre le deuxième est incrémenté de 1 et le dernier retombe à 0.
Si seulement quelques bogues ont été corrigés, sans changer du tout les fonctionnalités : le troisième nombre de la numérotation est incrémenté de 1, sans modifier les deux premiers nombres.
La page Wikipédia https://en.wikipedia.org/wiki/Software_versioning traite de ce sujet et propose d’autres règles.
Sous-étape b) Faire les mises à jour dans le code :
L’ajout de fonctionnalités ou la correction de bogues impliquent des modifications à apporter au code.
Sous-étape c) Documenter les mises à jour :
Les commentaires roxygen produisant les fiches d’aide doivent être mis à jour de façon à refléter les modifications apportées au code. Une correction d’un bogue ne nécessite pas toujours de mise à jour des fiches d’aide, mais un ajout de fonctionnalités en nécessite toujours.
Je conseille vivement aussi de documenter les mises à jour dans un fichier NEWS. Ce fichier est un point de repère pour un utilisateur d’un package qui souhaite identifier ce qui a changé lors d’une mise à jour, donc ce qui pourrait affecter son utilisation du package.
Il n’y a pas de consensus en R à propos de comment rédiger le fichier NEWS, ni de l’endroit où le placer dans les fichiers source. Nous pouvons observer, notamment, les pratiques suivantes :
- fichier
NEWSen format texte simple (dont le nom de fichier ne porte pas d’extension), placé dans le répertoire principal (au même niveau que les fichiersDESCRIPTIONetNAMESPACE); - fichier
NEWS.mden format Markdown, placé dans le répertoire principal; - fichier
NEWSen format texte simple (dont le nom de fichier ne porte pas d’extension), placé dans le sous-répertoireinst; - fichier
NEWS.Rden format.Rd, placé dans le sous-répertoireinst.
La première des pratiques est probablement la plus répandue.
Voici à quoi pourrait ressembler un fichier NEWS en format texte simple pour l’exemple du package manhattan, après l’avoir mis à jour.
Fichier "C:/coursR/manhattan/NEWS" :
Changements dans manhattan version 1.1.0 (2021-04-06)
* modifications des fonctions dist_manhattan et dist_manhattan_2 afin qu'elles
puissent calculer la distance entre plusieurs points
* ajout d'une methode plot pour un objet de classe "distman"
Changements dans manhattan version 1.0.0 (2021-03-29)
* premiere version du package manhattan
Nous pourrions également utiliser le commande usethis::use_news_md() pour produire un gabarit de fichier NEWS.md qu’il ne nous resterait plus qu’à éditer.
Si nous installions la version 1.1.0 du package, nous pourrions afficher son fichier NEWS avec les instructions suivantes :
news(package = "manhattan")Nous pouvons aussi voir le fichier NEWS des packages distribués sur le CRAN directement sur leur page web du CRAN (dans le champ Materials).
Sous-étape d) Revérifier et reconstruire le package
Il faut finalement construire et vérifier de nouveau le package. Un nouveau fichier compressé sera produit, portant le nom du package accompagné du nouveau numéro de version.
7 Résumé
Étapes de création d’un package R
1. Écrire les fonctions
Objets utilisables dans le corps des fonctions d’un package :
- arguments et variables locales,
- tout objet, public ou privé, contenu dans le package,
- objets provenant d’autres packages, à la condition d’inclure ces objets dans l’environnement des objets importés par le package,
- objets du package R de base.
Bonne pratique : Utiliser la forme <nomPackage>::<nomFonction>() pour appeler les fonctions provenant d’autres packages (sauf pour celles provenant du package base).
Si but = mettre sur le CRAN \(\rightarrow\) respecter http://cran.r-project.org/web/packages/policies.html
2. Créer l’arborescence de répertoires et fichiers du package
Répertoire principal, portant le nom du package, contenant :
- sous-répertoire nommé
R: répertoire des fichiers contenant le code source R des fonctions; - sous-répertoire nommé
man: répertoire des fichiers source des fiches d’aide; - sous-répertoire nommé
data: répertoire pour les jeux de données publics (au besoin); - fichier nommé
DESCRIPTION: informations descriptives générales du package (configurations); - fichier nommé
NAMESPACE: fichier permettant de définir quels objets sont publics (exportés) et d’identifier les fonctions d’autres packages (excluant le packagebase) utilisées dans le package (objets importés); - autres éléments, selon les besoins : fichier
NEWS, sous-répertoiresrc, etc.
Fonctions du package usethis utiles à cette étape :
create_package: création de l’arborescence de base, dans un projet RStudio;use_r: création de fichiers script R dans le sous-répertoire nomméR;use_data: ajout de jeux de données dans le sous-répertoire nommédata.
3. Écrire la documentation des fonctions et du package
Éléments à documenter :
- les fonctions publiques,
- les jeux de données,
- les classes et méthodes S4 ou RC publiques,
- les méthodes S3 pour des fonctions génériques (si pertinent),
- le package lui-même (recommandé).
Documentation d’un package avec roxygen2 :
Commentaires dans le code source R pour générer les fiches d’aide (contenu du sous-répertoire man) et le fichier NAMESPACE
- symbole de commentaire
roxygen:#'; - commentaires en entête des fonctions ou
- avant
"_PACKAGE"pour la fiche d’aide globale du package, - avant une chaîne de caractères contenant le nom d’un objet contenant des données;
- avant
- 1ère ligne \(\rightarrow\) titre de la fiche, suivi d’une ligne vide;
- 1er paragraphe \(\rightarrow\) champ
Descripionde la fiche, suivi d’une ligne vide; - autres paragraphes (optionnels) \(\rightarrow\) champ
Details; - tags :
@param: description des paramètres \(\rightarrow\) champArguments;@return: description de la sortie \(\rightarrow\) champValue;- pour le fichier
NAMESPACE:@export,@importFrom, etc.; - autres sections :
@examples,@author,@references,@format, etc.; - pour jumeler des fiches :
@describeIn,@rdname; - métadonnées :
@name,@aliases, etc.
Pour générer la documentation : devtools::document() ou devtools::check()
4. Vérifier et construire le package
Installations nécessaires :
- R,
- outils de développement de logiciel GNU,
- compilateur LaTeX (uniquement pour générer la documentation au format PDF).
R CMD check
Effectue diverses vérifications du package :
- option --as-cran avant de soumettre an CRAN,
- option --no-manual si aucun compilateur LaTeX n’est installé sur notre ordinateur.
R CMD build
Construit le package source (.tar.gz, = version Linux).
R CMD INSTALL --build
Construit le package binaire (.zip sous Windows, .tgz sous macOS )
Vérifier et construire le package avec le package devtools :
Fonctions du package devtools (à appeler dans la console) :
check: vérification, par défaut active l’option--as-cran;build: construction, par défaut crée le package source, version binaire obtenue avecbinary = TRUE;load_all: utile en cours de développement pour charger les fonctions dans la session;install: installation locale du package.
Vérifier et construire le package avec RStudio :
- Sous-étape a) Créer un projet RStudio avec notre package
- Sous-étape b) Configurer les options de RStudio
- Sous-étape c) Compiler à partir du menu « Build » de RStudio
- vérifier le package : menu « Build > Check Package »,
- créer le package source : menu « Build > Build Source Package »,
- créer le package binaire : menu « Build > Build Binary Package ».
5. Si désiré, partager le package
Il n’est pas compliqué de soumettre un package au CRAN : https://cran.r-project.org/submit.html
6. Au besoin, mettre à jour le package
Mise à jour = modification pour ajouter des fonctionnalités et/ou corriger des bogues :
- Sous-étape a) Incrémenter le numéro de version dans le fichier DESCRIPTION (et dans la fiche d’aide du package s’il y est mentionné)
- Sous-étape b) Faire les mises à jour dans le code
- Sous-étape c) Documenter les mises à jour :
- mettre à jour les commentaires
roxygenproduisant les fiches d’aide - décrire brièvement les changements dans fichier
NEWSouNEWS.md
(non obligatoire, mais c’est une bonne pratique)
- mettre à jour les commentaires
- Sous-étape d) Reconstruire et revérifier le package
Références
- R Core Team (2021). Writing R Extensions. R Foundation for Statistical Computing. Chapitres 1 et 2.
URL https://cran.r-project.org/doc/manuals/r-release/R-exts.html - Wickham, H. (2015). R packages. O’Reilly Media, Inc.
- URL première édition (quelques informations dans cette version sont déjà obsolètes) :
http://r-pkgs.had.co.nz/ - URL deuxième édition (en développement) : https://r-pkgs.org
- Bon chapitre résumé : https://r-pkgs.org/whole-game.html
- URL première édition (quelques informations dans cette version sont déjà obsolètes) :
Packages exploités dans ces notes :
- Wickham, H., Danenberg, P., Csárdi, G. et Eugster, M. (2020). roxygen2: In-Line Documentation for R. R package version 7.1.1. URL https://CRAN.R-project.org/package=roxygen2
- URL documentation en ligne https://roxygen2.r-lib.org/
- Wickham, H. et Bryan, J. (2021). usethis: Automate Package and Project Setup. R package version 2.0.1. URL https://CRAN.R-project.org/package=usethis
- URL documentation en ligne https://usethis.r-lib.org/
- Wickham, H., Hester, J. et Chang, W. (2020). devtools: Tools to Make Developing R Packages Easier.
R package version 2.3.2. https://CRAN.R-project.org/package=devtools
- URL documentation en ligne https://devtools.r-lib.org/
- feuille de triche https://rawgit.com/rstudio/cheatsheets/master/package-development.pdf
Tutoriel sur le web concernant le développement de packages R en utilisant les packages usethis et devtools :
- Bryan, J., Hester, J. et Wickham, H. (2019). Tutoriel présenté dans le cadre de la conférence UseR 2019 intitulé « Package Development ». URL https://github.com/jennybc/pkg-dev-tutorial
- Dray, M. (2019). Tutoriel web intitulé « Build an R package with {usethis} ». URL https://www.rostrum.blog/2019/11/01/usethis/
- Gelfand, S. (2019). Tutoriel web intitulé « usethis for reporting ». URL https://sharla.party/post/usethis-for-reporting/
- Meyer, C. et Hammerschmidt, D. (2020). Tutoriel web intitulé « How to write your own R package and publish it on CRAN ». URL https://www.mzes.uni-mannheim.de/socialsciencedatalab/article/r-package/
- Pileggi, S. (2020). Tutoriel web intitulé « Your first R package in 1 hour ». URL https://www.pipinghotdata.com/posts/2020-10-25-your-first-r-package-in-1-hour/
Pour le développement avec RStudio :
Annexe
Résolution de problèmes déjà rencontrés
Voici des problèmes déjà rencontrés par des étudiants du cours lors de la construction ou la vérification d’un package avec RStudio et leurs solutions.
Erreur « Installation failed »
PROBLÈME : Une erreur de ce type est produite par l’outil « Check Package » :
* checking whether package 'manhattan' can be installed ... ERROR
Installation failed.
See 'C:/coursR/NomRépertoireAvecAccents/manhattan.Rcheck/00install.out' for details.CAUSE : Le nom complet du répertoire du package contient des accents ou des espaces. (Cela ne cause pas toujours une erreur, ça dépend de la version de R utilisée et de votre système d’exploitation.)
SOLUTION : Utiliser un répertoire dont le nom complet ne comprend aucun accent, ni aucun espace.
Erreur de permission d’écriture dans un répertoire d’installation
PROBLÈME : Une erreur de ce type est produite par l’outil « Build Binary Package », « Install and Restart » ou « Clean and Rebuild » :
ERREUR\ : pas de permission pour installer dans le répertoire
'C:/Program Files/R/R-4.0.3/library'CAUSE : Le compte utilisé n’a pas la permission d’écrire dans le répertoire où R cherche à installer le package. Si vous travaillez à partir d’un compte administrateur, vous ne devriez pas rencontrer ce problème.
SOLUTION : Modifier les options des outils de construction pour demander l’installation dans un autre répertoire, un pour lequel le compte utilisateur à une permission en écriture.
Étape 1 : trouver le bon répertoire à utiliser
Il faut utiliser un des répertoires dans lesquels R recherche des installations de packages. Un vecteur contenant tous ces répertoires est retourné par la commande suivante dans la console R :
.libPaths()## [1] "C:/Users/Sophie/Documents/R/win-library/4.0"
## [2] "C:/Program Files/R/R-4.0.3/library"Dans mon cas, le premier répertoire énuméré, soit "C:/Users/Sophie/Documents/R/win-library/4.0", est localisé dans les fichiers du compte utilisateur. J’y ai donc assurément une permission en écriture. C’est lui que je vais utiliser.
Étape 2 : ajouter une option aux outils de construction
- ouvrir la fenêtre de configuration des outils de construction
- ajouter l’option
--library="C:/Users/Sophie/Documents/R/win-library/4.0"(à adapter selon le nom du répertoire que vous avez choisi d’utiliser) à deux endroits :
- la configuration de l’outil « Install and Restart » et « Clean and Rebuild » se fait dans le champ, nommé « Build and Reload - R CMD INSTALL additionnal options: »
- la configuration de l’outil « Build Binary Package » se fait dans le dernier champ, nommé « Build Binary Package - R CMD INSTALL additionnal options: »
Vérification qui gèle à l’étape « checking PDF version of manual »
PROBLÈME : Outil « Check Package » qui n’arrive pas à terminer son check, qui gèle à l’étape « checking PDF version of manual ».
CAUSE : Je ne sais pas exactement… Ça ressemble à un bogue.
SOLUTION : Modifier les options de l’outil « Check Package »
- ouvrir la fenêtre de configuration des outils de construction
- ajouter dans le champ nommé « Check Package - R CMD check additionnal options: » l’option suivante :
--no-manual