Calculs statistiques en R
Sophie Baillargeon, Université Laval
2021-02-24
Note préliminaire : Lors de leur dernière mise à jour, ces notes ont été révisées en utilisant R version 4.0.3 et le package broom version 0.7.4. Pour d’autres versions, les informations peuvent différer.
R est un environnement spécialisé dans les calculs statistiques. Voyons comment réaliser de tels calculs en R, qu’il s’agisse de production de statistiques descriptives, de manipulation des distributions de probabilité, de génération de nombres pseudo-aléatoires, de réalisation de tests ou d’ajustement de modèles.
1 Statistiques descriptives
Les fonctions permettant de calculer des statistiques descriptives en R sont très nombreuses. Les principales sont présentées ici, mais il en existe d’autres. Le tableau suivant classe les fonctions mentionnées ci-dessous selon le type de mesure calculée (mesure de position, de tendance centrale, de dispersion ou de fréquences) et selon le type de fonctionnement de la fonction (calcul vectoriel, données combinées en une ou plusieurs valeurs).
| Calcul | opère de façon vectorielle | combine, retourne une valeur | combine, retourne valeur(s) | combine, retourne un vecteur |
|---|---|---|---|---|
| mesure de position | pmin, pmax |
min, max, which.min, which.max |
range, quantile, summary |
cummin, cummax, rank |
| tendance centrale | mean, median |
summary |
||
| dispersion | sd, IQR, mad |
var, cov, cor |
||
| fréquences | table, ftable, xtabs, summary |
1.1 Fonctions retournant une seule statistique
Certaines fonctions de calcul de statistiques descriptives retournent en sortie une seule valeur. C’est le cas des fonctions suivantes :
- mesures de position :
min,max; - mesures de tendance centrale :
mean,median; - mesure de dispersion :
sd(écart-type),IQR(écart interquartile),mad(écart absolu médian).
Utilisons le jeu de données cars du package datasets pour présenter quelques exemples. Ce jeu de données contient 50 observations de 2 variables numériques.
str(cars)## 'data.frame': 50 obs. of 2 variables:
## $ speed: num 4 4 7 7 8 9 10 10 10 11 ...
## $ dist : num 2 10 4 22 16 10 18 26 34 17 ...# Moyenne des observations de la variable dist
mean(cars$dist)## [1] 42.98Si l’objet en entrée a plus d’une dimension, la sortie est tout de même de longueur 1. Donc tous les éléments contenus dans l’objet sont mis en commun pour faire le calcul.
max(cars)## [1] 120Fonctions which.max et which.min
Les fonctions min et max retournent respectivement la valeur la plus petite et la valeur la plus grande parmi les éléments d’un objet. Les fonctions which.max et which.min retournent pour leur part la position dans l’objet du premier maximum ou minimum.
which.min(cars$speed)## [1] 1Dans l’exemple précédent, il y a en fait deux observations qui prennent la valeur minimum de min(cars$speed). La commande suivante permet de trouver la position de toutes les observations prenant la valeur minimale.
which(cars$speed == min(cars$speed))## [1] 1 21.1.1 Traitement des données manquantes et argument na.rm
Les fonctions min, max, mean, median et sd, ainsi que quelques autres fonctions vues dans ces notes, ont un argument en commun nommé na.rm. Cet argument sert à indiquer à la fonction comment agir en présence de données manquantes (NA). Par défaut, na.rm prend la valeur FALSE pour ces fonctions. Cette valeur signifie que les données manquantes ne doivent pas être retirées avant d’effectuer le calcul. Cependant, en présence de données manquantes, ces fonctions ne sont pas en mesure de calculer des statistiques. Par exemple, supposons que nous voulions calculer la médiane des données dans le vecteur suivant.
x <- c(3, 6, NA, 8, 11, 15, 23)Si nous ne retirons pas la donnée manquante, nous obtenons le résultat suivant.
median(x)## [1] NACe résultat s’explique par le fait que la valeur de la médiane dépend de toutes les observations, incluant l’observation manquante, qui est inconnue. La valeur de la médiane est donc elle aussi inconnue. Pour calculer plutôt la médiane des observations non manquantes, il faut donner la valeur TRUE à l’argument na.rm comme suit.
median(x, na.rm = TRUE)## [1] 9.5Notons que la fonction na.omit permet de retirer les observations manquantes d’un objet R. Si l’objet est un vecteur, les éléments contenant NA sont retirés.
na.omit(x)## [1] 3 6 8 11 15 23
## attr(,"na.action")
## [1] 3
## attr(,"class")
## [1] "omit"La fonction na.omit ajoute deux attributs à l’objet, dont un pour identifier les observations retirées.
Remarquons que les deux commandes suivantes retournent le même résultat.
median(x, na.rm = TRUE)## [1] 9.5median(na.omit(x))## [1] 9.5Si la fonction na.omit reçoit en entrée une matrice ou un data frame, elle retire toutes les lignes contenant au moins un NA, comme dans cet exemple :
ex_jeu <- data.frame(x, y = c(2, NA, 8, 9, 6, NA, 2));
ex_jeu## x y
## 1 3 2
## 2 6 NA
## 3 NA 8
## 4 8 9
## 5 11 6
## 6 15 NA
## 7 23 2na.omit(ex_jeu)## x y
## 1 3 2
## 4 8 9
## 5 11 6
## 7 23 21.2 Fonctions pouvant retourner plusieurs statistiques
D’autres fonctions peuvent retourner plus d’une statistique, notamment les fonctions suivantes :
- mesures de position :
range,quantile; - résumé comprenant plusieurs mesures :
summary; - variances, covariances et corrélations :
var,cov,cor.
Fonctions range et quantile
La fonction range retourne à la fois le minimum est le maximum, comme dans cet exemple :
range(cars$speed)## [1] 4 25Une façon simple d’obtenir l’étendue d’observations à partir de la sortie de la fonction range est de procéder comme suit :
diff(range(cars$speed))## [1] 21La fonction quantile calcule des quantiles empiriques. Par défaut, elle retourne le minimum, le maximum et les quartiles, comme dans cet exemple :
quantile(cars$speed)## 0% 25% 50% 75% 100%
## 4 12 15 19 25L’argument probs permet de demander n’importe quels quantiles. Dans l’exemple suivant, les premiers et neuvièmes déciles sont demandés.
quantile(cars$speed, probs = c(0.1, 0.9))## 10% 90%
## 8.9 23.1Remarque : Il existe plusieurs façons de calculer des quantiles. La fonction quantile implémente 9 algorithmes de calcul de quantiles (voir help(quantile)).
Fonction summary
La fonction summary retourne les statistiques suivantes selon l’entrée qu’elle reçoit :
- vecteur numérique : minimum, premier quartile, médiane, moyenne, troisième quartile, maximum;
- facteur : fréquences des modalités (comme la fonction
tablevue plus loin); - matrice ou data frame : la fonction
summaryest appliquée séparément à chacune des colonnes.
Utilisons le jeu de données Puromycin du package datasets pour présenter quelques exemples d’utilisation de la fonction summary. Ce jeu de données contient 23 observations de 3 variables, dont deux numériques et une catégorique, stockée sous forme de facteur.
str(Puromycin)## 'data.frame': 23 obs. of 3 variables:
## $ conc : num 0.02 0.02 0.06 0.06 0.11 0.11 0.22 0.22 0.56 0.56 ...
## $ rate : num 76 47 97 107 123 139 159 152 191 201 ...
## $ state: Factor w/ 2 levels "treated","untreated": 1 1 1 1 1 1 1 1 1 1 ...
## - attr(*, "reference")= chr "A1.3, p. 269"# Vecteur numérique en entrée :
summary(Puromycin$rate)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 47.0 91.5 124.0 126.8 158.5 207.0# Facteur en entrée :
summary(Puromycin$state)## treated untreated
## 12 11# Data frame entier en entrée :
summary(Puromycin)## conc rate state
## Min. :0.0200 Min. : 47.0 treated :12
## 1st Qu.:0.0600 1st Qu.: 91.5 untreated:11
## Median :0.1100 Median :124.0
## Mean :0.3122 Mean :126.8
## 3rd Qu.:0.5600 3rd Qu.:158.5
## Max. :1.1000 Max. :207.0Fonctions var, cov et cor
La fonction var peut prendre en entrée un vecteur ou un objet à deux dimensions. Si elle reçoit en entrée un vecteur, elle calcule la variance empirique de toutes les valeurs, comme dans cet exemple :
var(cars$speed)## [1] 27.95918Cependant, si elle reçoit en entrée une matrice ou un data frame de valeurs numériques, elle considère que chaque colonne contient les observations d’une variable aléatoire. Elle va calculer une matrice de variances-covariances, comme dans cet exemple :
var(cars)## speed dist
## speed 27.95918 109.9469
## dist 109.94694 664.0608La fonction cov fait exactement le même calcul par défaut.
cov(cars)## speed dist
## speed 27.95918 109.9469
## dist 109.94694 664.0608Elle peut cependant calculer des covariances de Kendall ou de Spearman (toutes deux des statistiques non paramétriques basées sur les rangs des observations) au lieu de la covariance classique de Pearson. La fonction cor calcule des corrélations plutôt que des covariances. Elle aussi peut utiliser les définitions de Pearson (par défaut), Kendall et Spearman. Voici un exemple de calcul de matrice de corrélations de Spearman.
cor(cars, method = "spearman")## speed dist
## speed 1.0000000 0.8303568
## dist 0.8303568 1.00000001.3 Fonctions retournant un vecteur de statistiques
Certaines fonctions, telles que les suivantes, retournent autant de statistiques qu’il y a d’éléments dans l’objet donné en entrée.
- mesures de position :
cummin,cummax,pmin,pmax; - rangs :
rank.
Fonctions cummin et cummax
Les fonctions cummin et cummax calculent les minimums et les maximums cumulatifs. Comme nous pouvons le constater dans l’exemple suivant, la valeur en position i du vecteur retourné par une de ces deux fonctions est la valeur minimale ou maximale dans le sous-vecteur x[1:i].
cummin(x = c(-2, 4, -3, 4, 7, -6, 0))## [1] -2 -2 -3 -3 -3 -6 -6Fonctions pmin et pmax
Les fonctions pmin et pmax calculent le minimum et le maximum par position, entre autant de vecteurs que désiré, comme dans l’exemple suivant.
pmax(
c(-2, 4, -3, 4, 7, -6, 0),
c( 1, 2, 3, 4, 5, 6, 7),
c( 5, 0, -2, 4, 5, 3, 3)
)## [1] 5 4 3 4 7 6 7Ces fonctions sont utiles pour remplacer des valeurs par un seuil. Par exemple, l’instruction suivante permet de remplacer par zéro toute valeur négative contenue dans le vecteur en entrée.
pmax(c(-2, 4, -3, 4, 7, -6, 0), 0)## [1] 0 4 0 4 7 0 0Fonction rank
Certains tests statistiques non paramétriques utilisent des statistiques basées sur les rangs des observations. Voici un exemple d’obtention de ces rangs avec la fonction rank.
rank(c(-2, 4, -3, 4, 7, -6, 0))## [1] 3.0 5.5 2.0 5.5 7.0 1.0 4.0Par défaut, en cas d’égalité, le rang moyen est utilisé. Pour changer cette option, il faut modifier la valeur de l’argument ties.method. Dans l’exemple suivant, le rang minimum est retourné en cas d’égalité.
rank(c(-2, 4, -3, 4, 7, -6, 0), ties.method = "min")## [1] 3 5 2 5 7 1 41.4 Calcul de fréquences
Les fonctions table, xtabs et ftable permettent de calculer des fréquences.
Voici un petit jeu de données pour illustrer l’utilisation de ces fonctions. Il contient des observations concernant 7 individus fictifs : la couleur de leurs yeux, la couleur de leurs cheveux et leur genre.
sondage <- data.frame(
yeux = c("brun", "brun", "bleu", "brun", "vert", "brun", "bleu" ),
cheveux = c("brun", "noir", "blond", "brun", "brun", "blond", "brun" ),
genre = c("féminin", "masculin", "féminin", "féminin", "masculin", "féminin", "masculin")
)
sondage## yeux cheveux genre
## 1 brun brun féminin
## 2 brun noir masculin
## 3 bleu blond féminin
## 4 brun brun féminin
## 5 vert brun masculin
## 6 brun blond féminin
## 7 bleu brun masculinFonctions table
La fonction table permet de compter le nombre d’occurrences de chacune des modalités d’une variable catégorique dans des données. Demandons, par exemple, à table de compter le nombre d’individus dans les données sondage classés dans chacune des catégories de couleurs de cheveux.
table(sondage$cheveux)##
## blond brun noir
## 2 4 1La fonction table produit un tableau de fréquences à une variable si elle reçoit les observations d’une seule variable. Elle peut aussi produire des tableaux de fréquences croisées à deux variables ou plus.
# Exemple de tableau de fréquences à deux variables (avec variables nommées)
table(yeux = sondage$yeux, cheveux = sondage$cheveux)## cheveux
## yeux blond brun noir
## bleu 1 1 0
## brun 1 2 1
## vert 0 1 0# Exemple de tableau de fréquences à trois variables (data frame en entrée à table)
t3 <- table(sondage)
t3## , , genre = féminin
##
## cheveux
## yeux blond brun noir
## bleu 1 0 0
## brun 1 2 0
## vert 0 0 0
##
## , , genre = masculin
##
## cheveux
## yeux blond brun noir
## bleu 0 1 0
## brun 0 0 1
## vert 0 1 0Fonctions ftable
La fonction ftable retourne un tableau de fréquences sous la forme d’une table « plate » (en anglais flat, d’où le f dans le nom de la fonction) dans le cas d’un croisement de 3 variables ou plus, plutôt que sous la forme d’un array comme le fait la fonction table. Elle accepte les mêmes types d’entrées que table (série d’objets atomiques à une dimension ou objet récursif dont les éléments sont interprétables en facteurs) et peut aussi recevoir une sortie de la fonction table, comme dans l’exemple suivant.
ftable(t3)## genre féminin masculin
## yeux cheveux
## bleu blond 1 0
## brun 0 1
## noir 0 0
## brun blond 1 0
## brun 2 0
## noir 0 1
## vert blond 0 0
## brun 0 1
## noir 0 0Fonctions xtabs
La fonction xtabs fait le même calcul que les fonctions précédentes, mais elle prend en entrée une formule. Le tableau de fréquences à deux variables créé précédemment peut être réobtenu de la façon suivante avec xtabs.
xtabs(~ yeux + cheveux, data = sondage)## cheveux
## yeux blond brun noir
## bleu 1 1 0
## brun 1 2 1
## vert 0 1 0La fonction xtabs est utile lorsque les données que nous avons en main contiennent déjà des fréquences, car il est possible d’inclure une variable réponse contenant des dénombrements dans la formule que nous lui fournissons en entrée. Par exemple, xtabs permet de facilement retrouver le tableau de fréquences marginales croisées entre les variables yeux et cheveux à partir du tableau de fréquences à trois variables produit précédemment mis sous forme de data frame, qui a l’allure suivante.
t3_df <- as.data.frame(t3)
t3_df## yeux cheveux genre Freq
## 1 bleu blond féminin 1
## 2 brun blond féminin 1
## 3 vert blond féminin 0
## 4 bleu brun féminin 0
## 5 brun brun féminin 2
## 6 vert brun féminin 0
## 7 bleu noir féminin 0
## 8 brun noir féminin 0
## 9 vert noir féminin 0
## 10 bleu blond masculin 0
## 11 brun blond masculin 0
## 12 vert blond masculin 0
## 13 bleu brun masculin 1
## 14 brun brun masculin 0
## 15 vert brun masculin 1
## 16 bleu noir masculin 0
## 17 brun noir masculin 1
## 18 vert noir masculin 0Il suffit de procéder comme suit :
t2 <- xtabs(Freq ~ yeux + cheveux, data = t3_df)
t2## cheveux
## yeux blond brun noir
## bleu 1 1 0
## brun 1 2 1
## vert 0 1 0Autres fonctions relatives au calcul de fréquences
Les fonctions marginSums, addmargins et proportions permettent de calculer des fréquences marginales ou relatives à partir d’un tableau de fréquences. Voici quelques exemples d’utilisation de ces fonctions exploitant le tableau de fréquences à deux variables produit ci-dessus.
# Fréquences marginales en colonnes :
marginSums(t2, margin = 2)## cheveux
## blond brun noir
## 2 4 1# Fréquences marginales ajoutées au tableau :
addmargins(t2)## cheveux
## yeux blond brun noir Sum
## bleu 1 1 0 2
## brun 1 2 1 4
## vert 0 1 0 1
## Sum 2 4 1 7# Fréquences relatives croisées :
proportions(t2)## cheveux
## yeux blond brun noir
## bleu 0.1428571 0.1428571 0.0000000
## brun 0.1428571 0.2857143 0.1428571
## vert 0.0000000 0.1428571 0.0000000# Fréquences relatives conditionnelles à la variable yeux :
proportions(t2, margin = 1)## cheveux
## yeux blond brun noir
## bleu 0.50 0.50 0.00
## brun 0.25 0.50 0.25
## vert 0.00 1.00 0.00Transformation du format d’un objet de classe "table"
Les fonctions table et xtabs attribuent à l’objet qu’ils retournent en sortie la classe "table", comme nous pouvons le constater en observant l’objet t2.
attributes(t2)## $dim
## [1] 3 3
##
## $dimnames
## $dimnames$yeux
## [1] "bleu" "brun" "vert"
##
## $dimnames$cheveux
## [1] "blond" "brun" "noir"
##
##
## $class
## [1] "xtabs" "table"
##
## $call
## xtabs(formula = Freq ~ yeux + cheveux, data = t3_df)str(t2)## 'xtabs' int [1:3, 1:3] 1 1 0 1 2 1 0 1 0
## - attr(*, "dimnames")=List of 2
## ..$ yeux : chr [1:3] "bleu" "brun" "vert"
## ..$ cheveux: chr [1:3] "blond" "brun" "noir"
## - attr(*, "call")= language xtabs(formula = Freq ~ yeux + cheveux, data = t3_df)Il est parfois utile de transformer un objet de classe "table" en un array (matrice si la table croise deux variables) ou un data frame. Pour la transformation en array, il suffit de retirer l’attribut class avec la fonction unclass, comme dans cet exemple :
str(unclass(t2))## int [1:3, 1:3] 1 1 0 1 2 1 0 1 0
## - attr(*, "dimnames")=List of 2
## ..$ yeux : chr [1:3] "bleu" "brun" "vert"
## ..$ cheveux: chr [1:3] "blond" "brun" "noir"
## - attr(*, "call")= language xtabs(formula = Freq ~ yeux + cheveux, data = t3_df)Comme nous avons pu le constater dans un exemple précédent, la transformation en data frame crée pour sa part un jeu de données contenant une ligne par combinaison distincte des niveaux des facteurs croisés dans la table. Le data frame obtenu comporte une colonne par facteur, ainsi qu’une colonne nommée Freq contenant les fréquences dans la table. En voici un exemple :
str(as.data.frame(t2))## 'data.frame': 9 obs. of 3 variables:
## $ yeux : Factor w/ 3 levels "bleu","brun",..: 1 2 3 1 2 3 1 2 3
## $ cheveux: Factor w/ 3 levels "blond","brun",..: 1 1 1 2 2 2 3 3 3
## $ Freq : int 1 1 0 1 2 1 0 1 01.5 Énumération de combinaisons
Un fonction utile pour énumérer toutes les combinaisons des niveaux d’un facteur est expand.grid. Par exemple, retrouvons avec cette fonction toutes les combinaisons présentes dans le data frame t3_df créé précédemment.
expand.grid(
yeux = c("bleu", "brun", "vert"),
cheveux = c("blond", "brun", "noir"),
genre = c("féminin", "masculin")
)## yeux cheveux genre
## 1 bleu blond féminin
## 2 brun blond féminin
## 3 vert blond féminin
## 4 bleu brun féminin
## 5 brun brun féminin
## 6 vert brun féminin
## 7 bleu noir féminin
## 8 brun noir féminin
## 9 vert noir féminin
## 10 bleu blond masculin
## 11 brun blond masculin
## 12 vert blond masculin
## 13 bleu brun masculin
## 14 brun brun masculin
## 15 vert brun masculin
## 16 bleu noir masculin
## 17 brun noir masculin
## 18 vert noir masculinIl faut fournir en entrée à expand.grid les valeurs à combiner. Si les vecteurs ou facteurs contenant ces valeurs sont fournis avec des noms, comme dans l’exemple précédent (via les assignations), les colonnes du data frame retourné en sortie par expand.grid porteront ces noms.
Une autre fonction R permet d’énumérer des combinaisons possibles : la fonction combn. Il s’agit de combinaisons au sens mathématique cette fois, donc « de dispositions non ordonnées d’un certain nombre d’éléments d’un ensemble » (définition tirée du site web alloprof). Par exemple, voici toutes les combinaisons possibles de 3 éléments parmi l’ensemble c("Ève", "Jean", "Mia", "Paul"), trouvées par la fonction combn.
combn(x = c("Ève", "Jean", "Mia", "Paul"), m = 3)## [,1] [,2] [,3] [,4]
## [1,] "Ève" "Ève" "Ève" "Jean"
## [2,] "Jean" "Jean" "Mia" "Mia"
## [3,] "Mia" "Paul" "Paul" "Paul"Contrairement à expand.grid qui présente les combinaisons possibles ligne par ligne, chaque colonne de la sortie produite par combn représente une combinaison possible.
Notons finalement que la fonction choose mentionnée précédemment permet de compter le nombre de combinaisons possibles de k éléments d’un ensemble de taille n. Elle calcule donc le coefficient binomial (nk).
# Nombre de combinaisons possibles dans l'exemple précédent :
choose(n = 4, k = 3)## [1] 42 Distributions de probabilité
Le package stats de l’installation de base de R comprend, pour plusieurs distributions de probabilité, des fonctions de calcul de :
- la fonction de densité (forme
d*où*change selon la distribution), - la fonction de répartition (forme
p*) et - la fonction quantile (forme
q*).
La fiche d’aide ouverte par la commande help(Distributions) énumère toutes les distributions de probabilité offertes dans le package stats. Il existe aussi des fonctions relatives à d’autres distributions de probabilité dans des packages sur le CRAN (voir https://CRAN.R-project.org/view=Distributions pour découvrir ce qui est offert).
Ces fonctions sont utiles notamment pour calculer des valeurs critiques ou des seuils observés de tests d’hypothèses. Un exemple est présenté plus loin.
2.1 Fonction de densité
Les fonctions R implémentant des fonctions de densité ont un nom qui débute par le lettre d pour density. Leur premier argument est toujours un vecteur de valeurs en lesquelles calculer la fonction de densité. Les arguments suivants servent à spécifier les valeurs des paramètres de la distribution.
Dans le cas d’une variable aléatoire discrète, la fonction de densité est plus justement appelée fonction de masse. Il s’agit alors d’une probabilité, pour une variable aléatoire suivant une certaine distribution, de prendre une certaine valeur.
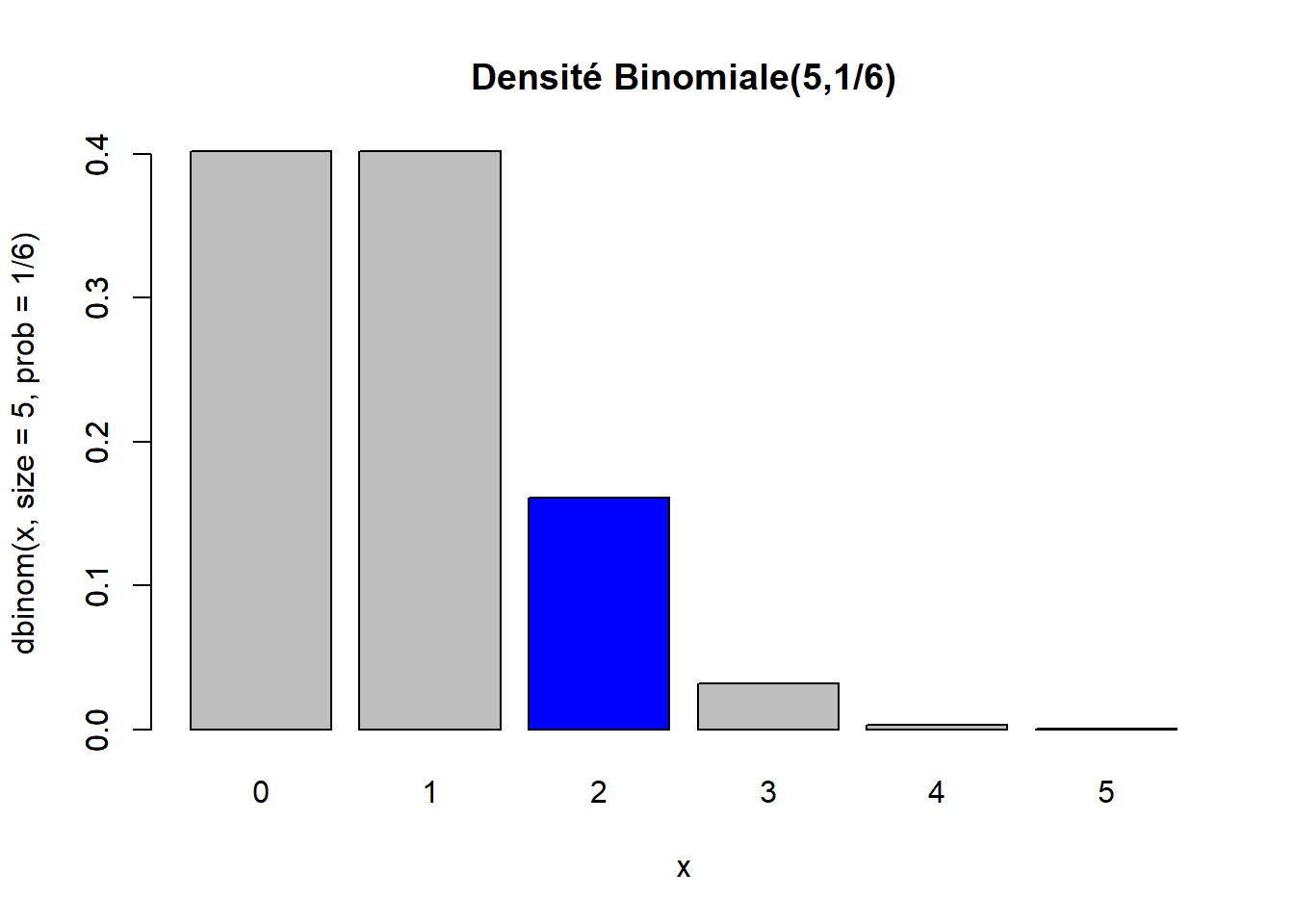
Exemple : distribution binomiale
Soit X une variable aléatoire représentant le nombre de 6 obtenus lors de 5 lancés d’un dé. Cette variable aléatoire suit une distribution binomiale de paramètres n=5 et p=1/6, donc X∼Bin(n=5,p=1/6).
Calculons P(X=2), soit la probabilité que la variable aléatoire X prenne la valeur 2.
dbinom(x = 2, size = 5, prob = 1/6)## [1] 0.160751La fonction dbinom implémente donc la fonction de masse d’une distribution binomiale. Les arguments size et prob sont les paramètres n et p de la distribution, selon la notation utilisée ci-dessus.
Les fonctions de la famille d* peuvent calculer plusieurs valeurs de densité par un seul appel de la fonction, car celles-ci acceptent des valeurs d’arguments de longueur supérieure à 1. Ces fonctions travaillent donc de façon vectorielle, comme presque toutes les fonctions de calcul en R.
Voici un exemple de code R permettant de représenter graphiquement la densité Bin(n=5,p=1/6) complète, en mettant en évidence la valeur calculée ci-dessus, soit P(X=2). (Nous verrons dans les notes sur les graphiques comment produire ce genre de figure et les suivantes.)
barplot(
height = dbinom(0:5, size = 5, prob = 1/6),
names.arg = 0:5,
main = "Densité Binomiale(5,1/6)",
xlab = "x",
ylab = "dbinom(x, size = 5, prob = 1/6)"
)
barplot(
height = c(0, 0, dbinom(2, size = 5, prob = 1/6), 0, 0, 0),
col = "blue",
add = TRUE
)
Exemple : distribution normale standard
Supposons maintenant que X est une variable aléatoire continue de distribution normale standard, donc X∼N(μ=0,σ2=1). La valeur de la fonction de densité pour cette distribution en la valeur x=1, souvent notée fX(1), vaut :
dnorm(x = 1)## [1] 0.2419707Voici une représentation graphique de la densité complète dans laquelle la valeur calculée ci-dessus est mise en évidence.
curve(expr = dnorm, xlim = c(-3, 3), main = "Densité N(0,1)")
segments(
x0 = c(-4, 1), y0 = dnorm(1), x1 = 1, y1 = c(dnorm(1), -1),
lty = 2, col = "blue"
)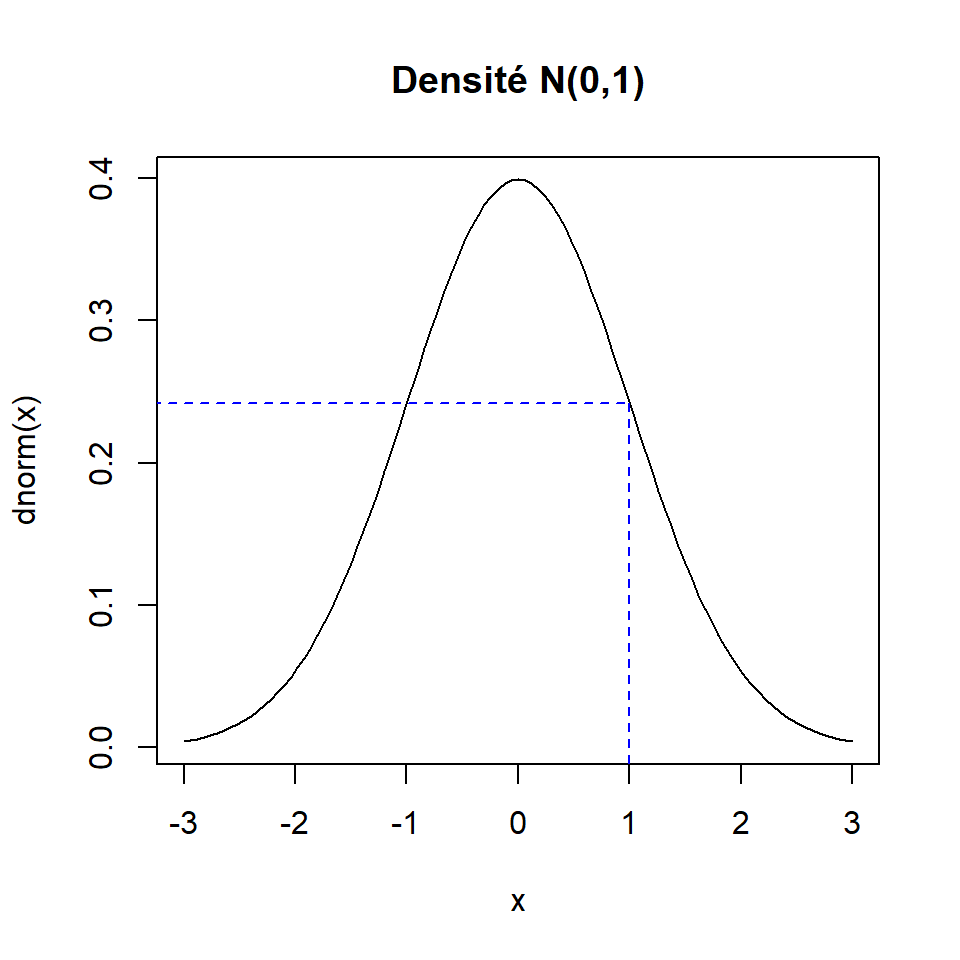
Ici, nous n’avons pas eu besoin de fournir des valeurs aux arguments de la fonction dnorm relatifs aux paramètres de la distribution, parce que nous avons utilisés leurs valeurs par défaut. Ces paramètres, pour la densité N(μ,σ2), sont représentés par les arguments mean = μ et sd = σ (remarquez que l’argument de la fonction R représente l’écart-type, pas la variance).
Tout comme le premier argument, nommé x, les arguments des fonctions de la famille d* représentant des paramètres de la distribution acceptent aussi en entrée plus d’une valeur. Voici un exemple, qui permet de calculer la densité en x=1 pour X∼N(μ=−2,σ2=1), X∼N(μ=0,σ2=2.25) et X∼N(μ=1,σ2=4) en un seul appel à la fonction dnorm.
dnorm(x = 1, mean = c(-2, 0, 1), sd = c(1, 1.5, 2))## [1] 0.004431848 0.212965337 0.1994711402.2 Fonction de répartition
La fonction de répartition d’une variable aléatoire X est définie par FX(x)=P(X≤x). Il s’agit donc toujours d’une probabilité, d’où le p au début des noms des fonctions R implémentant des fonctions de répartition.
Exemple : distribution normale standard
Prenons encore comme exemple la distribution normale standard. Nous avons donc X∼N(0,1). Calculons la valeur de la fonction de répartition de cette variable aléatoire en x=1.
pnorm(q = 1)## [1] 0.8413447Il s’agit de la valeur de la probabilité P(X≤1).
Le premier argument des fonctions de la famille p* ne se nomme pas x, il se nomme plutôt q. Cette lettre réfère au mot quantile et souligne le lien entre les fonctions de répartition et les fonctions quantiles. Les arguments suivants des fonctions de la famille p* permettent de spécifier les valeurs des paramètres de la distribution, comme pour les fonctions de la famille p*.
Voici une illustration graphique du lien entre la fonction de densité et la fonction de répartition en utilisant la distribution normale standard. La valeur de la fonction de répartition en un point quelconque x (dans le graphique x=1) est égale à l’aire sous la courbe de densité entre −∞ et x.
par_default <- par(mfrow = c(1, 2))
curve(expr = dnorm, xlim = c(-3, 3), main = "Densité N(0,1)")
x <- seq(from = -3, to = 1, length = 100)
polygon(x = c(x, 1, -3), y = c(dnorm(x), 0, 0), col = "blue")
curve(
expr = pnorm, xlim = c(-3, 3),
main = "Fonction de répartition N(0,1)", xname = "q"
)
segments(
x0 = c(-4, 1), y0 = pnorm(1), x1 = 1, y1 = c(pnorm(1), -1),
lty = 2, col = "blue"
)
par(par_default)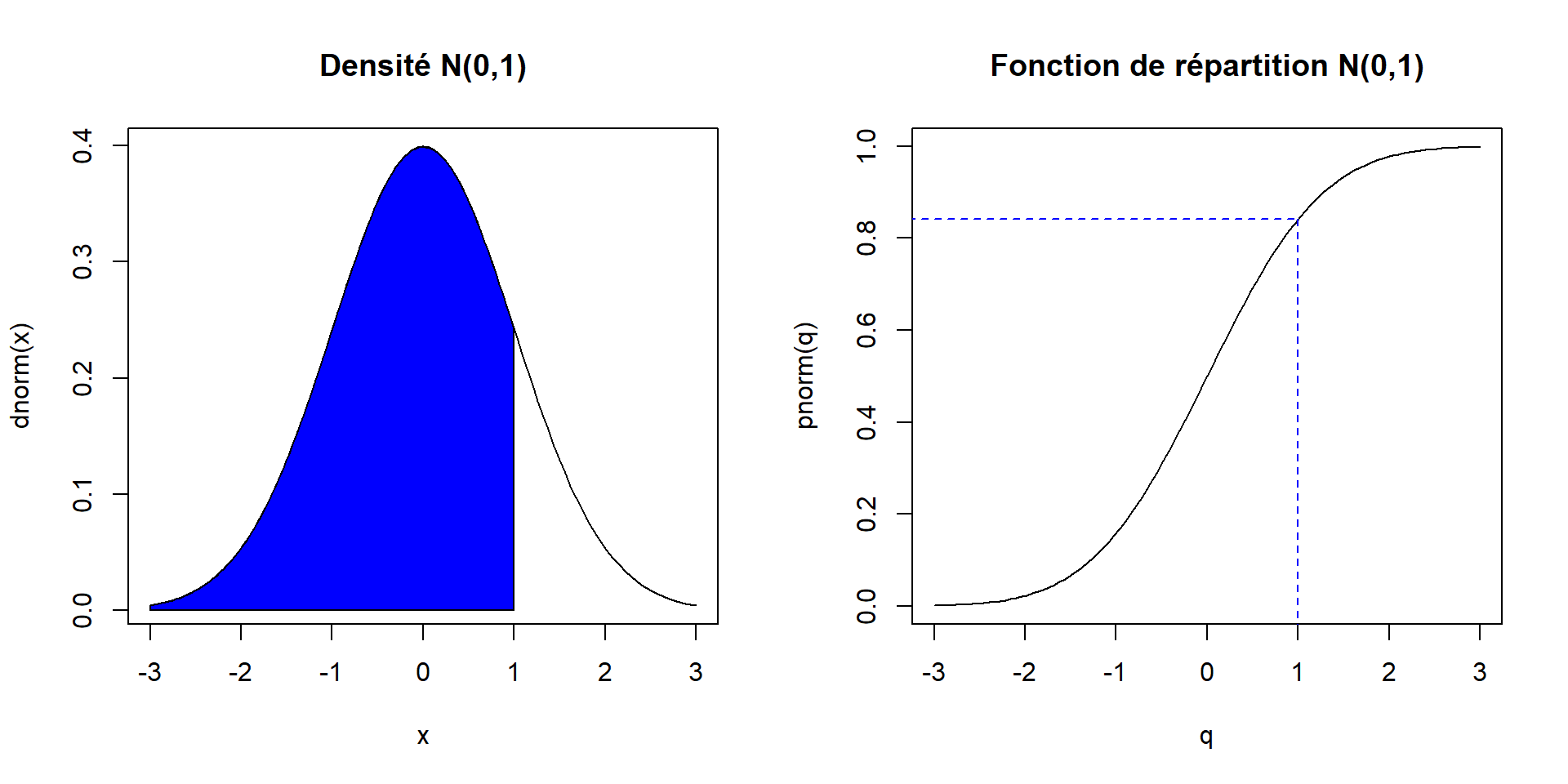
2.3 Fonction quantile
La fonction quantile est l’inverse généralisé de la fonction de répartition. Les fonctions R implémentant des fonctions quantile ont un nom qui débute par le lettre q pour quantile.
Exemple : distribution normale standard
Pour clore l’exemple de la distribution normale standard, voyons de quoi à l’air la fonction quantile de cette distribution.
Premièrement, calculons la valeur de la fonction quantile en un point, disons en p=0.8413447.
qnorm(p = 0.8413447)## [1] 0.9999998Il s’agit de la valeur x pour laquelle P(X≤x)=0.8413447, où X∼N(0,1).
Le premier argument d’une fonction de la famille q* se nomme p. Cette notation peut nous aider à nous rappeler que cet argument représente une probabilité et accepte donc seulement des valeurs entre 0 et 1. Encore une fois, les arguments suivants des fonctions de la famille q* permettent de spécifier les valeurs des paramètres de la distribution.
Le graphique suivant illustre le lien entre la fonction de répartition et la fonction quantile. Le graphique de la fonction quantile est simplement obtenu en inversant les axes du graphique de la fonction de répartition.
par_default <- par(mfrow = c(1,2), pty = "s")
curve(
expr = pnorm, xlim = c(-3, 3), n = 1000, ylim = c(0, 1), asp = 6,
main = "Fonction de répartition N(0,1)", xname = "q"
)
segments(
x0 = c(-4, 1), y0 = pnorm(1), x1 = 1, y1 = c(pnorm(1), -1),
lty = 2, col = "blue"
)
curve(
expr = qnorm, xlim = c(0, 1), n = 1000, ylim = c(-3, 3), asp = 1/6,
main = "Fonction quantile N(0,1)", xname = "p"
)
segments(
x0 = c(-0.2, pnorm(1)), y0 = qnorm(pnorm(1)),
x1 = pnorm(1), y1 = c(qnorm(pnorm(1)), -4),
lty = 2, col = "blue"
)
par(par_default)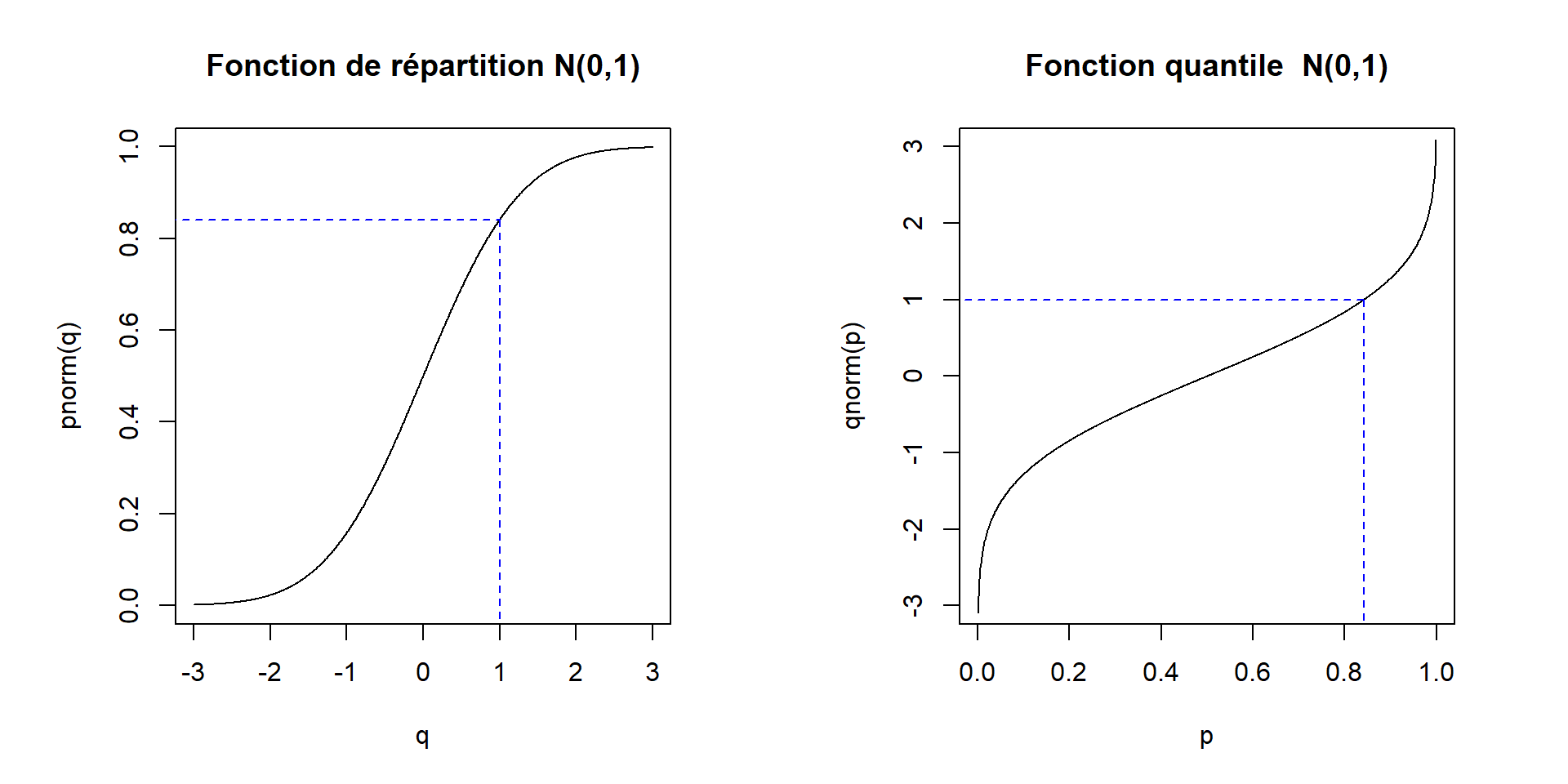
3 Génération de nombres pseudo-aléatoires
En R, les fonctions r* permettent de générer pseudo-aléatoirement des observations selon une certaine distribution désignée par *. La lettre r au début de leur nom signifie random.
Par exemple, voici la représentation graphique de 3 échantillons générés aléatoirement selon 3 distributions différentes : des distributions normale, uniforme continue et khi-deux. Pour chaque échantillon, nous traçons l’histogramme des observations simulées pour représenter leur densité empirique. Nous superposons à cet histogramme la courbe de densité de la distribution théorique à partir de laquelle les observations ont été générées.
par_default <- par(mfrow = c(1, 3))
# Densité normale d'espérance 5 et de variance 4
x1 <- rnorm(1000, mean = 5, sd = 2)
hist(x = x1, freq = FALSE, ylab = "Proportion", main = "Densité empirique de x1")
curve(expr = dnorm(x, mean = 5, sd = 2), add = TRUE, col = "blue")
title(main = "courbe bleue = densité visée N(5,4)", line = 0.5)
# Densité uniforme continue entre 2 et 8
x2 <- runif(1000, min = 2, max = 8)
hist(x = x2, freq = FALSE, ylab = "Proportion", main = "Densité empirique de x2")
curve(expr = dunif(x, min = 2, max = 8), add = TRUE, col = "blue")
title(main = "courbe bleue = densité visée U(2,8)", line = 0.5)
# Densité chi-carré à 4 degrés de liberté
x3 <- rchisq(1000, df = 4)
hist(x = x3, freq = FALSE, ylab = "Proportion", main = "Densité empirique de x3")
curve(expr = dchisq(x, df = 4), add = TRUE, col = "blue")
title(main = expression(paste("courbe bleue = densité visée ", chi[4]^2)), line = 0.5)
par(par_default)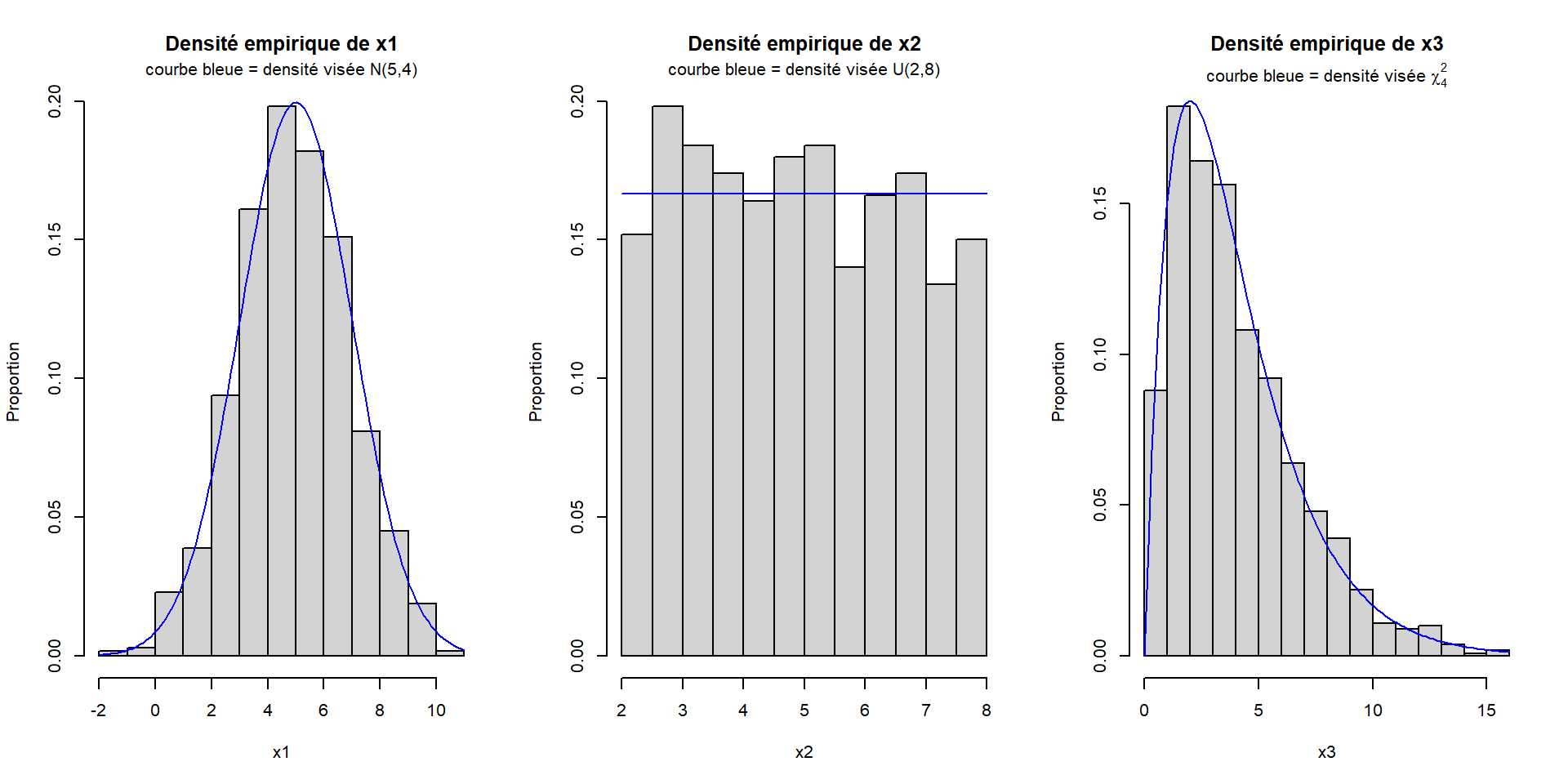
Comme nous pouvons le constater sur ces graphiques, la distribution empirique des observations générées avec ces fonctions se rapproche vraiment de la distribution théorique demandée. En générant un nombre encore plus grand d’observations (ici nous en avons généré 1000 pour chaque distribution), la densité empirique se rapprocherait encore plus de la densité théorique.
3.1 Fonction sample
La fonction sample permet de tirer un échantillon aléatoire parmi un ensemble d’éléments fourni à son argument x sous forme de vecteur.
sample(x = c("Luc", "Kim", "Paul", "Ève"))## [1] "Kim" "Luc" "Ève" "Paul"Ce vecteur peut contenir des données de n’importe quel type.
sample(x = 1:6)## [1] 1 3 2 6 5 4Par défaut, sample effectue un tirage sans remise et sélectionne autant d’éléments que l’ensemble de départ en contient. La fonction effectue donc une permutation aléatoire des éléments de l’ensemble de départ. L’argument size permet cependant de contrôler la taille de l’échantillon tiré.
sample(x = 1:6, size = 5)## [1] 2 4 3 5 6L’argument replace permet quant à lui de spécifier si le tirage doit être effectué avec (replace = TRUE) ou sans remise (replace = FALSE, valeur par défaut).
sample(x = 1:6, size = 10, replace = TRUE)## [1] 4 2 4 3 2 2 1 5 6 1Il est aussi possible d’attribuer des probabilité de sélection à chacun des éléments grâce à l’argument prob. Par défaut, tous les éléments ont une probabilité égale d’être tiré.
sample(x = 1:6, size = 20, replace = TRUE, prob = c(1/2, rep(1/10, times = 5)))## [1] 1 2 1 3 4 1 1 1 4 3 1 6 1 2 1 1 1 4 6 2La fonction sample.int est très similaire à la fonction sample, mais elle prend comme premier argument un seul entier, n, et tire aléatoirement size entiers entre 1 et n.
sample.int(n = 6, size = 4)## [1] 2 4 1 5Si nous souhaitons sélectionner aléatoirement des observations (lignes) dans un jeu de données, il est d’usage de sélectionner d’abord des entiers compris entre 1 et le nombre total d’observations, puis d’extraire du jeu de donnée les observations sur les lignes portant les numéros sélectionnés. Voici un exemple, utilisant célèbres données iris, incluses dans l’installation de base de R dans le data frame nommé iris (du package datasets).
str(iris)## 'data.frame': 150 obs. of 5 variables:
## $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
## $ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
## $ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
## $ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
## $ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...index_ech <- sample.int(n = nrow(iris), size = 5, replace = FALSE)
index_ech## [1] 136 18 19 69 91iris_ech <- iris[index_ech, ]
iris_ech## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 136 7.7 3.0 6.1 2.3 virginica
## 18 5.1 3.5 1.4 0.3 setosa
## 19 5.7 3.8 1.7 0.3 setosa
## 69 6.2 2.2 4.5 1.5 versicolor
## 91 5.5 2.6 4.4 1.2 versicolor3.2 Germe de la génération pseudo-aléatoire
Les nombres générés avec les fonctions r* et les échantillons tirés avec sample sont qualifiés de pseudo-aléatoires, car ils proviennent d’un algorithme déterministe qui tente de reproduire le hasard. Un tel algorithme est nommé en anglais random number generator (RNG) ou pseudo-random number generator. La fiche d’aide ouverte par la commande help(Random) contient de l’information sur la génération de nombres pseudo-aléatoires en R.
Plusieurs RNG sont implémentés en R. Celui utilisé par défaut, nommé Mersenne Twister, a été choisi parce qu’il est réputé être bon. Sans entrer dans les détails du fonctionnement des RNG implémentés dans le package base de R, il faut savoir qu’ils travaillent tous à partir d’une séquence de nombres appelée germe (en anglais seed). En contrôlant ce germe, il est possible de générer de nouveau, autant de fois que désiré, les mêmes valeurs.
Par défaut, R contrôle le germe des RNG de façon automatique. Chaque fois qu’une commande faisant intervenir un RNG est évaluée, R crée un nouveau germe à partir, notamment, de l’heure à laquelle la commande est soumise. Ainsi, deux générations pseudo-aléatoires consécutives ne produisent en général pas le même résultat.
sample(x = letters, size = 5)## [1] "e" "k" "v" "n" "u"sample(x = letters, size = 5)## [1] "k" "p" "u" "o" "n"En tout temps, il est possible de connaître le germe du RNG en R. Il est stocké dans un objet nommé .Random.seed. Cet objet est un vecteur numérique de longueur 626 pour le RNG Mersenne Twister. Voyons de quoi ont l’air les premiers éléments de ce vecteur à différents moments.
str(.Random.seed)## int [1:626] 10403 76 722238004 552337818 -1279533595 -1491617409 -434728698 -1712133..sample(x = letters, size = 5)## [1] "o" "t" "l" "z" "b"str(.Random.seed)## int [1:626] 10403 82 722238004 552337818 -1279533595 -1491617409 -434728698 -1712133..sample(x = letters, size = 5)## [1] "z" "e" "b" "u" "a"str(.Random.seed)## int [1:626] 10403 87 722238004 552337818 -1279533595 -1491617409 -434728698 -1712133..Nous constatons qu’au moins un élément de ce vecteur (le deuxième élément) change à chaque fois que nous appelons la fonction sample. La fonction set.seed permet de fixer le germe du RNG à partir d’une seule valeur entière. Par exemple, la commande suivante :
set.seed(753)spécifie le germe suivant :
str(.Random.seed)## int [1:626] 10403 624 302719261 615841082 -1828406925 -1314498536 214287161 14262109..En soumettant de nouveau le même appel à la fonction sample, nous obtenons l’échantillon suivant.
sample(x = letters, size = 5)## [1] "v" "r" "g" "i" "t"La soumission de cette commande a eu pour effet de modifier le germe.
str(.Random.seed)## int [1:626] 10403 6 47777699 -819670847 -326033232 523235278 1163109177 1104276299 1..Donc si nous resoumettons la commande sample, nous n’obtiendrons sûrement pas le même résultat.
sample(x = letters, size = 5)## [1] "l" "q" "f" "e" "k"Par contre, si nous fixons de nouveau le germe à partir de l’entier 753 avec set.seed, nous arrivons à obtenir de nouveau l’avant-dernier échantillon généré.
set.seed(753)
sample(x = letters, size = 5)## [1] "v" "r" "g" "i" "t"À n’importe quel moment, dans n’importe qu’elle session R, nous obtenons l’échantillon {v, r, g, i, t} si nous soumettons la commande set.seed(753) avant la commande sample(letters, size = 5).
L’utilité de fixer le germe d’une génération pseudo-aléatoire est de rendre son résultat reproductible.
4 Tests statistiques
Il existe des fonctions R pour faire des tests statistiques de base. En voici quelques-unes :
- tests sur une ou des moyennes :
t.test; - tests sur une ou des proportions :
prop.test,binom.test; - tests de comparaison de variances :
var.test,bartlett.test; - tests sur une corrélation :
cor.test; - tests pour une distribution :
shapiro.test,ks.test; - tests non paramétriques :
wilcox.test,kruskal.test,friedman.test; - tests sur des fréquences : méthode
summarypour un objet de classetable(produit par la fonctiontableouxtabs),chisq.test,fisher.test,mantelhaen.test.
Utilisons encore une fois les données iris pour construire un exemple de test de comparaison de moyennes. Nous allons comparer les largeurs moyennes des sépales des espèces versicolor et virginica.
Exemple : test t bilatéral de comparaison de moyennes, avec variances inégales
t.test(
formula = Sepal.Width ~ Species,
data = iris,
subset = Species %in% c("versicolor", "virginica")
)##
## Welch Two Sample t-test
##
## data: Sepal.Width by Species
## t = -3.2058, df = 97.927, p-value = 0.001819
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -0.33028364 -0.07771636
## sample estimates:
## mean in group versicolor mean in group virginica
## 2.770 2.974Lorsque nous connaissons la valeur d’une statistique de test ainsi que sa loi sous l’hypothèse nulle, il est possible de calculer en R le seuil observé du test avec la bonne fonction p*. Par exemple, dans le test t ci-dessus, nous pouvons retrouver la valeur du seuil observé ainsi :
# multiplication par 2, car le test est bilatéral et la loi t est symétrique
pt(q = -3.2058, df = 97.927)*2 ## [1] 0.001819258Exemple : test non paramétrique équivalent
wilcox.test(
formula = Sepal.Width ~ Species,
data = iris,
subset = Species %in% c("versicolor", "virginica"),
exact = FALSE
)##
## Wilcoxon rank sum test with continuity correction
##
## data: Sepal.Width by Species
## W = 841, p-value = 0.004572
## alternative hypothesis: true location shift is not equal to 05 Ajustement de modèles
Il existe plusieurs fonctions en R pour ajuster des modèles. Les modèles les plus usuels sont :
- modèles linéaires, dont la régression :
lmpour linear model; - modèle d’analyse de la variance :
lmouaov; - modèles linéaires généralisés :
glm; - modèles linéaires mixtes (peuvent contenir des effets aléatoires) :
lmerdu packagelme4oulmedu packagenlme; - modèles non linéaires :
nls.
À titre d’exemples, ajustons deux modèles sur les données iris.
Exemple : régression linéaire simple entre la largeur et la longueur des sépales
reg <- lm(formula = Sepal.Width ~ Sepal.Length, data = iris)
reg##
## Call:
## lm(formula = Sepal.Width ~ Sepal.Length, data = iris)
##
## Coefficients:
## (Intercept) Sepal.Length
## 3.41895 -0.06188Exemple : ANOVA pour comparer les largeurs de sépales moyennes entre toutes les espèces
ANOVA <- aov(formula = Sepal.Width ~ Species, data = iris)
ANOVA## Call:
## aov(formula = Sepal.Width ~ Species, data = iris)
##
## Terms:
## Species Residuals
## Sum of Squares 11.34493 16.96200
## Deg. of Freedom 2 147
##
## Residual standard error: 0.3396877
## Estimated effects may be unbalanced5.1 Formules
Ces fonctions d’ajustement de modèles prennent obligatoirement en entrée une formule R assignée à l’argument formula, tout comme la fonction xtabs. D’autres fonctions acceptent aussi en entrée une formule, sans que ce type d’argument soit obligatoire. C’est le cas des fonctions aggregate, ftable, plusieurs des fonctions effectuant un test (p. ex. t.test, wilcox.test) et certaines fonctions graphiques (p. ex. plot, boxplot).
Les fonctions prenant une formule en entrée ont toujours un argument data pour spécifier d’où tirer les variables incluses dans la formule.
Une formule s’écrit sous la forme y ~ x1 + x2 où y représente la variable réponse (ou à expliquer, ou dépendante, ou endogène) et x1 et x2 des variables explicatives (ou indépendantes, ou exogènes). Dans la partie de droite, les opérateurs suivants peuvent apparaître :
- «
+» : pour ajouter des termes; - «
-» : pour soustraire des termes; - «
0» : ou1pour représenter l’ordonnée à l’origine (par défaut tout modèle linéaire comporte une ordonnée à l’origine, pour la retirer il faut ajouter- 1ou+ 0à la partie de droite de la formule); - «
.» : pour représenter toutes les variables dans le jeu de données fournit en argumentdata, autres que la variable mise à la gauche du~(note : le point signifie autre chose dans la fonctionupdate).
Opérateurs propres aux facteurs :
- «
:» : pour les termes d’interaction entre facteurs; - «
*» : pour le croisement de facteurs (x1*x2est équivalent àx1 + x2 + x1:x2); - «
^» : pour le croisement de facteurs jusqu’à un certain niveau d’interaction (par exemple(x1 + x2 + x3)^2va inclure tous les termes de croisement des facteurs jusqu’aux interactions doublesx1 + x2 + x3 + x1:x2 + x1:x3 + x2:x3, mais pas l’interaction triplex1:x2:x3); - «
%in%» : pour les facteurs emboîtés (dansx2 %in% x1,x2est emboîté dansx1).
Autre opérateur :
- «
|» : (p. ex.y ~ x1 | x2) n’a pas toujours exactement la même signification selon la fonction, il représente parfois :
Les formules peuvent inclurent des appels à des fonctions pour transformer les variables. Par exemple, pour ajuster un modèle sur la racine carrée de la variable réponse, nous pouvons écrire sqrt(y) ~ x1 + x2. Certaines transformations pourraient faire intervenir un ou des opérateurs ayant une signification modifiée dans la partie de droite d’une formule. Par exemple, imaginons que nous voulons ajuster un modèle avec une seule variable explicative créée en additionnant les valeurs des variables x1 et x2. La formule y ~ x1 + x2 n’ajuste pas ce modèle puisque dans la partie de droite d’une formule l’opérateur + signifie « ajouter des termes » et non plus « additionner des valeurs ». Alors est-il possible d’ajuster le modèle souhaité (sans ajouter une nouvelle variable dans les données)?
Oui, c’est possible grâce à la fonction I. Il faut encadrer l’opération arithmétique à effectuer dans la formule d’un appel à la fonction I. Par exemple, y ~ I(x1 + x2) ajuste un modèle à une seule variable explicative formée de la somme des valeurs de x1 et x2. Ainsi, I() permet d’utiliser la signification usuelle des opérateurs et non celle spécifique aux formules.
5.1.1 Exemples
Voici quelques exemples, dans lesquels nous omettrons de nommer le premier argument, soit formula, pour alléger la présentation et parce qu’il est usuel de ne pas nommer ce premier argument.
5.1.1.1 Retrait de l’ordonnée à l’origine
reg <- lm(Sepal.Width ~ Sepal.Length - 1, data = iris)
# ou encore :
reg <- lm(Sepal.Width ~ Sepal.Length + 0, data = iris)coef(summary(reg))## Estimate Std. Error t value Pr(>|t|)
## Sepal.Length 0.5117739 0.008939966 57.24562 2.422615e-1035.1.1.2 Régression log-log
reg <- lm(log(Sepal.Width) ~ log(Sepal.Length), data = iris)coef(summary(reg))## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.3057236 0.14573780 8.959402 1.293792e-15
## log(Sepal.Length) -0.1129573 0.08275742 -1.364920 1.743495e-015.1.1.3 Régression polynomiale
reg <- lm(Sepal.Width ~ Sepal.Length + Sepal.Length^2, data = iris)coef(summary(reg))## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 3.4189468 0.25356227 13.483658 1.552431e-27
## Sepal.Length -0.0618848 0.04296699 -1.440287 1.518983e-01Non, ça n’a pas fonctionné. Dans une formule, l’opérateur ^ ne signifie pas exposant. Pour demander à R d’utiliser la signification usuelle de l’opérateur, et non celle spécifique aux formules, il faut encadrer le terme contenant l’opérateur de I() comme suit :
reg <- lm(Sepal.Width ~ Sepal.Length + I(Sepal.Length^2), data = iris)coef(summary(reg))## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 6.41583572 1.58499197 4.047866 8.327686e-05
## Sepal.Length -1.08556027 0.53624556 -2.024372 4.474291e-02
## I(Sepal.Length^2) 0.08570656 0.04475502 1.915016 5.743267e-02
Création de variables catégoriques pour les exemples à venir (nous verrons dans les notes sur le prétraitement de données comment utiliser la fonction cut) :
iris$Sepal.Length_catego <- cut(
x = iris$Sepal.Length,
breaks = c(-Inf, quantile(iris$Sepal.Length, probs = c(1/3, 2/3)), Inf),
right = FALSE
)
iris$Petal.Width_catego <- cut(
x = iris$Petal.Width,
breaks = c(-Inf, quantile(iris$Petal.Width, probs = c(1/3, 2/3)), Inf),
right = FALSE
)5.1.1.4 Anova à 3 facteurs, modèle complet
ANOVA <- aov(Sepal.Width ~ Species*Sepal.Length_catego*Petal.Width_catego, data = iris)
# ou encore
ANOVA <- aov(
Sepal.Width ~ (Species + Sepal.Length_catego + Petal.Width_catego)^3,
data = iris
)summary(ANOVA)## Df Sum Sq Mean Sq F value Pr(>F)
## Species 2 11.345 5.672 64.620 < 2e-16 ***
## Sepal.Length_catego 2 3.843 1.922 21.891 5.53e-09 ***
## Petal.Width_catego 1 0.872 0.872 9.934 0.00199 **
## Species:Sepal.Length_catego 3 0.077 0.026 0.291 0.83194
## Species:Petal.Width_catego 1 0.006 0.006 0.073 0.78754
## Sepal.Length_catego:Petal.Width_catego 1 0.047 0.047 0.540 0.46357
## Species:Sepal.Length_catego:Petal.Width_catego 1 0.002 0.002 0.026 0.87212
## Residuals 138 12.114 0.088
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 15.1.1.5 Anova à 3 facteurs, modèle complet sans l’interaction triple
ANOVA <- aov(
Sepal.Width ~ Species*Sepal.Length_catego*Petal.Width_catego -
Species:Sepal.Length_catego:Petal.Width_catego,
data = iris
)
# ou encore
ANOVA <- aov(
Sepal.Width ~ (Species + Sepal.Length_catego + Petal.Width_catego)^2,
data = iris
)summary(ANOVA)## Df Sum Sq Mean Sq F value Pr(>F)
## Species 2 11.345 5.672 65.076 < 2e-16 ***
## Sepal.Length_catego 2 3.843 1.922 22.046 4.83e-09 ***
## Petal.Width_catego 1 0.872 0.872 10.004 0.00192 **
## Species:Sepal.Length_catego 3 0.077 0.026 0.293 0.83046
## Species:Petal.Width_catego 1 0.006 0.006 0.073 0.78681
## Sepal.Length_catego:Petal.Width_catego 1 0.047 0.047 0.544 0.46199
## Residuals 139 12.116 0.087
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 15.1.2 Arguments accompagnant les formules
Pour les fonctions prenant une formule comme premier argument, cet argument est souvent le seul argument obligatoire.
reg <- lm(iris$Sepal.Width ~ iris$Sepal.Length)
coef(summary(reg))## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 3.4189468 0.25356227 13.483658 1.552431e-27
## iris$Sepal.Length -0.0618848 0.04296699 -1.440287 1.518983e-01Cependant, un argument formula est toujours accompagné d’un argument data. Typiquement, l’utilisateur fournit à data un data frame contenant en colonnes les variables à inclure dans la formule. Utiliser l’argument data permet d’alléger la formule. Les noms de variables dans la formule sont d’abord recherchés parmi les noms des éléments de data.
reg <- lm(Sepal.Width ~ Sepal.Length, data = iris) # (plus de iris$ dans la formule)
coef(summary(reg))## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 3.4189468 0.25356227 13.483658 1.552431e-27
## Sepal.Length -0.0618848 0.04296699 -1.440287 1.518983e-01
En plus de l’argument data, les fonctions prenant une formule en entrée ont la plupart du temps les arguments :
subsetpour spécifier un sous-ensemble de données à utiliser (par défaut toutes les observations dedatasont utilisées) etna.actionpour spécifier quoi faire avec les valeurs manquantes (voirhelp(na.fail)).
5.1.2.1 Ajustement d’un modèle sur un sous-ensemble des données
Un argument subset se spécifie comme l’argument subset de la fonction subset. Sa valeur doit être un vecteur logique dont la longueur est égale au nombre d’observations dans le jeu de données fourni à l’argument data. Voici un exemple.
reg <- lm(Sepal.Width ~ Sepal.Length, data = iris, subset = Species == "setosa")
coef(summary(reg))## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -0.5694327 0.5217119 -1.091470 2.805148e-01
## Sepal.Length 0.7985283 0.1039651 7.680738 6.709843e-105.1.2.2 Modification du traitement des valeurs manquantes
iris_2 <- iris
iris_2$Sepal.Length[6] <- NA # pour insérer une donnée manquante (pas de NA dans iris)La valeur fournie à l’argument na.action est le nom d’une fonction qui traite les données manquantes dans un jeu de données. Le comportement par défaut de l’argument na.action est régi par l’option de session du même nom.
getOption("na.action")## [1] "na.omit"Dans mon cas, na.action prend donc par défaut la valeur na.omit, ce qui a pour conséquence que les observations avec au moins une valeur de variable manquante sont omises (ligne complète non considérée).
Nous pourrions fournir à l’argument une autre des fonctions listées dans la fiche d’aide help(na.fail). Par exemple, l’ajustement du modèle n’est pas effectué lorsque les données comportent des données manquantes si la valeur na.fail est fournie à na.action.
reg <- lm(Sepal.Width ~ Sepal.Length, data = iris_2, na.action = na.fail)## Error in na.fail.default(list(Sepal.Width = c(3.5, 3, 3.2, 3.1, 3.6, 3.9, :
## missing values in object5.2 Manipulation de la sortie
Lorsque nous affichons dans la console un objet produit en sortie d’une fonction d’ajustement de modèle, la sortie obtenue est brève.
reg <- lm(Sepal.Width ~ Sepal.Length, data = iris)
reg##
## Call:
## lm(formula = Sepal.Width ~ Sepal.Length, data = iris)
##
## Coefficients:
## (Intercept) Sepal.Length
## 3.41895 -0.06188En réalité, cet objet est une liste contenant plusieurs éléments.
str(reg, list.len = 5)## List of 12
## $ coefficients : Named num [1:2] 3.4189 -0.0619
## ..- attr(*, "names")= chr [1:2] "(Intercept)" "Sepal.Length"
## $ residuals : Named num [1:150] 0.3967 -0.1157 0.0719 -0.0343 0.4905 ...
## ..- attr(*, "names")= chr [1:150] "1" "2" "3" "4" ...
## $ effects : Named num [1:150] -37.4445 -0.6255 0.0564 -0.0485 0.471 ...
## ..- attr(*, "names")= chr [1:150] "(Intercept)" "Sepal.Length" "" "" ...
## $ rank : int 2
## $ fitted.values: Named num [1:150] 3.1 3.12 3.13 3.13 3.11 ...
## ..- attr(*, "names")= chr [1:150] "1" "2" "3" "4" ...
## [list output truncated]
## - attr(*, "class")= chr "lm"L’objet obtenu de la fonction d’ajustement de modèle ne s’affiche pas comme une liste parce qu’un attribut classe lui est attribué et que la fonction polymorphe print possède une définition spécifique aux objets de cette classe.
class(reg)## [1] "lm"print(reg)##
## Call:
## lm(formula = Sepal.Width ~ Sepal.Length, data = iris)
##
## Coefficients:
## (Intercept) Sepal.Length
## 3.41895 -0.06188C’est une caractéristique orientée objet du langage R. Dans la terminologie de R, une fonction polymorphe est appelée fonction générique et les différentes définitions de cette fonction sont appelées méthodes. Les fonctions génériques, telles que les fonctions print, plot et summary, ont un comportement qui varie en fonction de la classe du premier argument qui leur est fourni en entrée.
Rappelons que taper le nom d’un objet dans la console est en fait un raccourci pour soumettre la fonction print avec l’objet à afficher en argument.
Si nous retirons l’attribut classe de l’objet, nous retombons sur un affichage usuel pour un objet de type liste.
class(reg) <- NULL
reg # résultat non affiché, car trop longL’attribut classe peut même contenir plus d’une classe.
ANOVA <- aov(Sepal.Width ~ Species, data = iris)
class(ANOVA)## [1] "aov" "lm"Souvent, le nom de la classe d’un objet est le nom de la fonction qui a produit cet objet. Les objets retournés par la fonction aov ont deux classes, car en réalité la fonction aov appelle la fonction lm.
5.2.1 Fonctions génériques d’extraction d’information
Voici la liste des fonctions génériques les plus couramment utilisées pour tirer de l’information d’un objet produit par une fonction d’ajustement de modèle :
summary: pour afficher un résumé des informations plus long que ce qui est affiché avecprintet pour produire des résultats supplémentaires;coef(ou son aliascoefficients) etconfint: pour afficher les coefficients et pour produire des intervalles de confiance pour les coefficients d’un modèle;residualsetfitted: pour extraire les résidus et les valeurs prédites;predict: pour effectuer des prédictions pour de nouvelles observations;anova: pour calculer la table d’analyse de la variance (ANOVA) du modèle;model.tablesetTukeyHSD(pour la classeaov) : pour calculer les moyennes par niveaux de facteurs et pour faire des comparaisons multiples de Tukey sur ces moyennes;deviance,logLik,AIC,BIC: pour extraire la déviance, la log-vraisemblance maximisée, le AIC et le BIC.
L’utilisation de ces fonctions est la façon usuelle en R d’extraire des résultats relatifs à un modèle. Par exemple, pour extraire les coefficients d’un modèle, nous pouvons utiliser :
reg <- lm(Sepal.Width ~ Sepal.Length, data = iris)
coef(reg)## (Intercept) Sepal.Length
## 3.4189468 -0.0618848Notons cependant qu’il est aussi possible d’extraire cette information en accédant directement aux éléments de reg, qui est une liste.
reg$coefficients## (Intercept) Sepal.Length
## 3.4189468 -0.0618848
Voici quelques exemples d’utilisation des fonctions d’extraction d’information :
reg <- lm(Sepal.Width ~ Sepal.Length + Petal.Width, data = iris)
summary(reg)##
## Call:
## lm(formula = Sepal.Width ~ Sepal.Length + Petal.Width, data = iris)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.99563 -0.24690 -0.00503 0.23354 1.01131
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.92632 0.32094 6.002 1.45e-08 ***
## Sepal.Length 0.28929 0.06605 4.380 2.24e-05 ***
## Petal.Width -0.46641 0.07175 -6.501 1.17e-09 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.3841 on 147 degrees of freedom
## Multiple R-squared: 0.234, Adjusted R-squared: 0.2236
## F-statistic: 22.46 on 2 and 147 DF, p-value: 3.091e-09confint(reg)## 2.5 % 97.5 %
## (Intercept) 1.2920761 2.5605655
## Sepal.Length 0.1587655 0.4198079
## Petal.Width -0.6082076 -0.3246210str(residuals(reg))## Named num [1:150] 0.1916 -0.25054 0.00731 -0.06376 0.32053 ...
## - attr(*, "names")= chr [1:150] "1" "2" "3" "4" ...predict(
reg,
newdata = data.frame(
Sepal.Length = c(5, 6),
Petal.Width = c(1, 2)
)
)## 1 2
## 2.906340 2.729213ANOVA <- aov(Sepal.Width ~ Species + Sepal.Length_catego, data = iris)
summary(ANOVA)## Df Sum Sq Mean Sq F value Pr(>F)
## Species 2 11.345 5.672 62.70 < 2e-16 ***
## Sepal.Length_catego 2 3.843 1.922 21.24 8.13e-09 ***
## Residuals 145 13.119 0.090
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1anova(ANOVA)## Analysis of Variance Table
##
## Response: Sepal.Width
## Df Sum Sq Mean Sq F value Pr(>F)
## Species 2 11.3449 5.6725 62.697 < 2.2e-16 ***
## Sepal.Length_catego 2 3.8433 1.9216 21.240 8.129e-09 ***
## Residuals 145 13.1187 0.0905
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1model.tables(ANOVA, type = "means")## Tables of means
## Grand mean
##
## 3.057333
##
## Species
## setosa versicolor virginica
## 3.428 2.77 2.974
## rep 50.000 50.00 50.000
##
## Sepal.Length_catego
## [-Inf,5.4) [5.4,6.3) [6.3, Inf)
## 2.919 3.081 3.157
## rep 46.000 53.000 51.000TukeyHSD(ANOVA)## Tukey multiple comparisons of means
## 95% family-wise confidence level
##
## Fit: aov(formula = Sepal.Width ~ Species + Sepal.Length_catego, data = iris)
##
## $Species
## diff lwr upr p adj
## versicolor-setosa -0.658 -0.80045516 -0.5155448 0.0000000
## virginica-setosa -0.454 -0.59645516 -0.3115448 0.0000000
## virginica-versicolor 0.204 0.06154484 0.3464552 0.0025694
##
## $Sepal.Length_catego
## diff lwr upr p adj
## [5.4,6.3)-[-Inf,5.4) 0.16172436 0.01819230 0.3052564 0.0229762
## [6.3, Inf)-[-Inf,5.4) 0.23756010 0.09272626 0.3823939 0.0004549
## [6.3, Inf)-[5.4,6.3) 0.07583574 -0.06387887 0.2155503 0.40571675.2.2 Résultats additionnels fournis par summary
La fonction générique summary ne fait pas que produire un affichage du modèle ajusté plus complet que la fonction générique print. Elle produit des résultats supplémentaires concernant le modèle. Par exemple, pour un modèle produit avec lm, comparons ce que produit directement la fonction lm et ce que produit la fonction générique summary pour un objet retourné par lm.
reg <- lm(Sepal.Width ~ Sepal.Length, data = iris)
reg_summary <- summary(reg)
sort(names(reg))## [1] "assign" "call" "coefficients" "df.residual" "effects"
## [6] "fitted.values" "model" "qr" "rank" "residuals"
## [11] "terms" "xlevels"sort(names(reg_summary))## [1] "adj.r.squared" "aliased" "call" "coefficients" "cov.unscaled"
## [6] "df" "fstatistic" "r.squared" "residuals" "sigma"
## [11] "terms"Seulement 4 éléments de la liste reg portent des noms aussi présents dans la liste reg_summary. Et des éléments de même nom dans les deux listes ne contiennent pas toujours la même chose.
reg$coefficients## (Intercept) Sepal.Length
## 3.4189468 -0.0618848reg_summary$coefficients## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 3.4189468 0.25356227 13.483658 1.552431e-27
## Sepal.Length -0.0618848 0.04296699 -1.440287 1.518983e-01Les éléments produits par summary tendent à contenir plus de détails que les éléments obtenus directement de lm.
La fonction summary réalise donc des calculs supplémentaires relatifs au modèle. Par exemple, pour un modèle ajusté avec lm, elle réalise les tests sur les termes du modèle et calcule le coefficient de détermination (R2), soit une mesure souvent utilisée pour évaluer la qualité de la prédiction du modèle.
reg_summary$r.squared## [1] 0.01382265Pour un modèle d’analyse de la variance ajusté avec aov, la fonction summary produit la table d’ANOVA complète, tout comme la fonction anova le fait.
ANOVA <- aov(Sepal.Width ~ Species + Sepal.Length_catego, data = iris)
ANOVA_summary <- summary(ANOVA)
str(ANOVA_summary)## List of 1
## $ :Classes 'anova' and 'data.frame': 3 obs. of 5 variables:
## ..$ Df : num [1:3] 2 2 145
## ..$ Sum Sq : num [1:3] 11.34 3.84 13.12
## ..$ Mean Sq: num [1:3] 5.6725 1.9216 0.0905
## ..$ F value: num [1:3] 62.7 21.2 NA
## ..$ Pr(>F) : num [1:3] 2.40e-20 8.13e-09 NA
## - attr(*, "class")= chr [1:2] "summary.aov" "listof"5.2.3 Mise en forme avec le package broom
Le package broom offre des fonctions pour faciliter la manipulation de sorties d’une fonction d’ajustement de modèle. Les trois principales fonctions de ce package sont les suivantes :
tidy: produit un résumé des principaux résultats statistiques d’un modèle, dans le cas d’un modèle linéaire il s’agit d’une table des tests sur les coefficients du modèle;augment: ajoute aux données sur lesquelles le modèle a été ajusté des informations tirées du modèle, comme des résidus et des valeurs prédites;glance: réunit dans un seul data frame à une ligne plusieurs statistiques globales au modèle, comme des statistiques d’ajustement du modèle.
Voici quelques exemples :
reg <- lm(Sepal.Width ~ Sepal.Length, data = iris)library(broom)
tidy(reg) # très similaire à coef(summary(reg)) pour une sortie de lm## # A tibble: 2 x 5
## term estimate std.error statistic p.value
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 (Intercept) 3.42 0.254 13.5 1.55e-27
## 2 Sepal.Length -0.0619 0.0430 -1.44 1.52e- 1head(augment(reg))## # A tibble: 6 x 8
## Sepal.Width Sepal.Length .fitted .resid .hat .sigma .cooksd .std.resid
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 3.5 5.1 3.10 0.397 0.0121 0.435 0.00516 0.919
## 2 3 4.9 3.12 -0.116 0.0154 0.436 0.000563 -0.269
## 3 3.2 4.7 3.13 0.0719 0.0195 0.436 0.000277 0.167
## 4 3.1 4.6 3.13 -0.0343 0.0218 0.436 0.0000709 -0.0798
## 5 3.6 5 3.11 0.490 0.0136 0.434 0.00893 1.14
## 6 3.9 5.4 3.08 0.815 0.00859 0.431 0.0154 1.89glance(reg)## # A tibble: 1 x 12
## r.squared adj.r.squared sigma statistic p.value df logLik AIC BIC deviance df.residual
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int>
## 1 0.0138 0.00716 0.434 2.07 0.152 1 -86.7 179. 188. 27.9 148
## # ... with 1 more variable: nobs <int>Plus d’informations peuvent être trouvées dans la documentation du package : https://broom.tidymodels.org/
6 Résumé
Statistiques descriptives
| Calcul | opère de façon vectorielle | combine, retourne une valeur | combine, retourne valeur(s) | combine, retourne un vecteur |
|---|---|---|---|---|
| mesure de position | pmin, pmax |
min, max, which.min, which.max |
range, quantile, summary |
cummin, cummax, rank |
| tendance centrale | mean, median |
summary |
||
| dispersion | sd, IQR, mad |
var, cov, cor |
||
| fréquences | table, ftable, xtabs, summary |
- Traitement des données manquantes :
- argument
na.rm, - fonction
na.omit;
- argument
- calcul de fréquences marginales ou relatives à partir d’un tableau de fréquences :
marginSums,addmargins,proportions. - énumération de combinaisons :
expand.grid,combn.
Distributions de probabilité et génération de nombres aléatoires
| Famille de fonction | Description |
|---|---|
d* |
fonction de densité de la distribution * |
p* |
fonction de répartition de la distribution * |
q* |
fonction quantile de la distribution * |
r* |
génération pseudo-aléatoirement d’observations selon la distribution * |
Liste de toutes les distributions * offertes en R de base : help(Distributions)
- fonction
sample(ousample.int) : tirage aléatoire d’échantillons - fonction
set.seed: pour fixer le germe du tirage pseudo-aléatoire
Tests statistiques
Quelques fonctions R pour faire des tests statistiques de base :
- tests sur une ou des moyennes :
t.test; - tests sur une ou des proportions :
prop.test,binom.test; - tests de comparaison de variances :
var.test,bartlett.test; - tests sur une corrélation :
cor.test; - tests pour une distribution :
shapiro.test,ks.test; - tests non paramétriques :
wilcox.test,kruskal.test,friedman.test; - tests sur des fréquences : méthode
summarypour un objet de classetable(produit par la fonctiontableouxtabs),chisq.test,fisher.test,mantelhaen.test.
Ajustement de modèles
Quelques fonctions R pour ajuster des modèles :
Formules R
Écriture générale : y ~ x1 + x2
où y = variable réponse, x1 et x2 = variables explicatives.
Opérateurs acceptés et leur signification :
+pour ajouter des termes;-pour soustraire des termes;- 1ou+ 0pour retirer l’ordonnée à l’origine;.pour représenter toutes les variables dans le jeu de données, autres que la variable mise à la gauche du~;:pour les termes d’interaction entre facteurs;*pour le croisement de facteurs;^croisement de facteurs jusqu’à un certain niveau d’interaction;%in%pour les facteurs emboîtés;|pour un conditionnement ou spécifier des effets aléatoires
I() pour utiliser la signification usuelle des opérateurs et non celle spécifique aux formules.
Sortie d’une fonction d’ajustement de modèle
Affichage (print) vs liste contenant les objets retournés
La liste retournée a une ou des classes → programmation orientée objet
Fonctions pour tirer de l’information d’un objet produit par une fonction d’ajustement de modèle :
summary: plus de résultats relatifs au modèle + affichage d’un résumé des informations;coef(ou son aliascoefficients) etconfint: coefficients et leurs intervalles de confiance;residualsetfitted: résidus et valeurs prédites;predict: prédiction pour une nouvelle observation;anova: table d’analyse de la variance du modèle;model.tablesetTukeyHSD(pour la classeaov) : moyennes par niveaux de facteurs et comparaisons multiples de Tukey;deviance,logLik,AIC,BIC: déviance, log-vraisemblance maximisée, AIC et BIC.
Références
Les informations présentées dans ces notes proviennent des fiches d’aide du logiciel R :
- R Core Team (2020). R: A Language and Environment for Statistical Computing. R Foundation for Statistical Computing, Vienna, Austria. URL https://www.R-project.org/
Le manuel suivant de R contient aussi plusieurs informations concernant les calculs statistiques en R :
- R Core Team (2020). An Introduction to R. R version 4.0.3. URL https://cran.r-project.org/doc/manuals/r-release/R-intro.html
Documentation du package mentionné qui ne vient pas avec l’installation de base de R :
- package
broom:- page web du package sur le CRAN : https://CRAN.R-project.org/package=broom
- documentation du package : https://broom.tidymodels.org/
Une très grande quantité de méthodes statistiques sont implémentées en R. Voici quelques bonnes références sur le sujet.
- Livres :
- Hothorn, T. et Everitt, B.S. (2014). A Handbook of Statistical Analyses Using R, third edition. CRC Press.
- Millot, G. (2018). Comprendre et réaliser les tests statistiques à l’aide de R: manuel de biostatistique, 4e édition. De Boeck Supérieur.
- Sites web contenant plusieurs exemples d’analyses statistiques en R :
- https://stats.idre.ucla.edu/other/dae/ (contient aussi les mêmes exemples avec d’autres logiciels statistiques)
- https://www.statmethods.net/about/sitemap.html
- Ressources pour dénicher des packages R implémentant des méthodes statistiques particulières :
- Task views de R : http://cran.r-project.org/web/views/
- Dépôt informatique de packages R en bio-informatique : http://www.bioconductor.org/
Pour aller plus loin :
- Braun, W. J. et Murdoch, D. (2007). A First Course in Statistical Programming with R. Cambridge University Press.